
| Marcin Bójko | Piotr Krzyżanowski |
Przy tworzeniu kolejnych wersji biblioteki PETSc aktywnie uczestniczyli (i uczestniczą!) jej użytkownicy. Dlatego PETSc nie zapomina o wygodzie programisty i oferuje najrozmaitsze udogodnienia przydatne podczas uruchamiania aplikacji:
Pamiętajmy także, że ostateczną miarą rzeczywistej efektywności użytej metody rozwiązania danego problemu jest czas i koszt działania komputera plus czas i koszt pisania programu!
Często, badając nowe algorytmy dla równań cząstkowych chcielibyśmy przeprowadzić eksperymenty, porównujące nasz algorytm z innymi dostępnymi. Łatwo to osiągnąć używając PETSc, ze względu na mnogość zaimplementowanych solverów i łatwość dodawania własnych, oraz możliwości profilingu: możemy zbadać nie tylko liczbę iteracji, ale także wymagania pamięciowe, koszt obliczeń, itp. Co więcej, prowadząc eksperymenty możemy szybko weryfikować w praktyce pewne hipotezy.
Jednym z istniejących rozwiązań jest język Fortran 90 oraz tworzony obecnie standard HPF. Fortran 90 w części zwalnia użytkownika od myślenia o równoległości, albowiem to kompilator może decydować, kiedy urównoleglić jakąś prostą operację. Tak na przykład działa kompilator F90 na Connection Machine CM-200. Podobne podejście stosuje np. Cray, gdzie jest dostępny tzw. zrównoleglacz kodu, dopisujący w programie odpowiednie dyrektywy dla kompilatora, powodujące wykorzystanie przetwarzania wektorowego lub równoległego.
Innym rozwiązaniem, jak sądzimy, bardziej elastycznym, jest wykorzystanie popularnego standardu MPI (Message Passing Interface) razem z "klasycznymi" językami programowania, takimi jak C czy Fortran. Właśnie ten drugi wariant wybrali autorzy PETSc, opierając na nim swoją implementację. PETSc daje z jednej strony dużo stosunkowo łatwych w użyciu narzędzi pozwalających na programowanie algorytmów równoległych bez potrzeby zagłębiania się w detale komunikacji między procesorami, itp. Z drugiej zaś strony, użytkownik wciąż może (jeśli chce) odzyskać pełną kontrolę nad realizacją przetwarzania równoległego i używać bezpośrednio funkcji standardu MPI.
Wydaje się nam także, iż raczej nie warto używać PETSc do zadań algebry liniowej z macierzami "gęstymi" (nawet dużego wymiaru). Lepiej skorzystać bezpośrednio z optymalizowanych bibliotek numerycznych, zazwyczaj dostarczanych przez producenta razem z superkomputerem, albo korzystać wprost z BLASów lub procedur LAPACKa czy ScaLAPACKa.
W końcu, wymieńmy parę wad PETSc. Zazwyczaj użycie PETSc prowadzi do wygenerowania kodu wynikowego o sporej objętości. Jest to problem, ale z drugiej strony, nawet najmniejszy program w PETSc daje nam wciąż dostęp do jej funkcjonalności i np. licznych procedur wychwytujących błędy. Również szybkość działania niektórych procedur zostawia trochę do życzenia. Poważną wadą PETSc jest dość stroma krzywa uczenia się: nie sposób zacząć programować w PETSc bez zapoznania się z manualem, a ten z kolei nie jest niestety najłatwiejszą lekturą...
Niniejszy podręcznik ma na celu po pierwsze - zainteresowanie tym nowoczesnym i wygodnym pakietem szerszego grona studentów, a także osób zajmujących się obliczeniami naukowymi/eksperymentami numerycznymi w Polsce, a po drugie - oszczędzenie początkującemu użytkownikowi PETSc mozolnego przegryzania się przez oficjalny manual i man pages: ułatwienie wstępnego zapoznania się z pakietem i szybkie (w miarę bezbolesne) umożliwienie mu rozpoczęcia pisania własnych programów wykorzystujących PETSc.
Adresujemy go przede wszystkim do tych osób, które chcą prowadzić eksperymenty numeryczne w dziedzinie równań różniczkowych cząstkowych typu eliptycznego, liniowych bądź nieliniowych. Dlatego ograniczyliśmy się wyłącznie do wersji sekwencyjnej PETSc, kwestiom równoległości poświęcając jedynie kilka komentarzy i uwag. Mimo to i tak objętość niniejszego podręcznika dorównuje blisko połowie objętości oficjalnego manuala, choć dotyczy jedynie 20% jego zawartości. Szczególny nacisk położyliśmy na zilustrowanie omawianych funkcji możliwie licznymi przykładami praktycznych zastosowań. Nie wszystkie przykłady są programami dającymi się uruchomić - często dla większej przejrzystości kodu zrezygnowaliśmy z linii nie związanych bezpośrednio z omawianym zagadnieniem. W każdym rozdziale znajdują się przykłady podsumowujące i te dadzą się skompilować i uruchomić. Ponieważ ten podręcznik ma jedynie pomóc w rozpoczęciu pracy z PETSc, nie będą też prezentowane i omawiane techniki związane z optymalizacją kodu pod kątem obliczeń równoległych. Jak wcześniej argumentowaliśmy, nawet w wersji sekwencyjnej (jednoprocesorowej) pakiet jest bardzo przydatnym narzędziem.
Jeśli będzie takie zapotrzebowanie, być może napiszemy kiedyś drugą część podręcznika, poświęconą technikom równoległym. Będziemy także wdzięczni za wszystkie uwagi dotyczące niniejszej wersji podręcznika. Prosimy je kierować pocztą elektroniczną na adres przykry@mimuw.edu.pl. Będziemy je uwzględniać w miarę możliwości w wersji elektronicznej podręcznika.
Aby korzystać z PETSc niezbędna jest znajomość w stopniu średnim programowania w języku C (podstawowe funkcje, wskaźniki, tablice i struktury) na poziomie standardu ANSI C. W zasadzie znajomość standardu Kernighan-Ritchie [12] też w zupełności wystarczy.

Aby rozwiązać powyższy układ przy pomocy komputera (być może równoległego) należy:
Inicjatorem projektu jest Barry Smith, który w 1991 roku napisał pierwszą wersję pakietu do rozwiązywania układów równań liniowych: PETSc 1.0. W ciągu kilku lat zestaw procedur przekształcił się w duży pakiet narzędzi do rozwiązywania równań różniczkowych cząstkowych, używany w wielu renomowanych ośrodkach obliczeniowych na świecie. Obecnie nad PETSc pracują Satish Balay, Willam Gropp, Lois Curfman McInnes i Barry Smith z Argonne National Laboratory (USA), wspomagani okazjonalnie przez grupy studentów - praktykantów. Akronim PETSc oznacza Portable, Extensible Toolkit for Scientific computation. Jak już wspomnieliśmy, pakiet jest w dużym stopniu niezależny od platformy sprzętowej. Programy można kompilować i uruchamiać na różnych architekturach, nie tracąc przy tym zbyt wiele z efektywności kodu, a jednocześnie wykorzystując potencjał konkretnego komputera. Obecnie obsługiwane architektury to:
Drugim przymiotnikiem z nazwy jest Extensibile (rozszerzalny) - autorzy położyli duży nacisk na to aby pakiet był modularny i można było także wykorzystywać, ostatnio bardzo modne, programowanie obiektowe. "PETSc jest dowodem na to, że nowoczesne metody programowania mogą ułatwić rozwój wielkich aplikacji numerycznych w Fortranie, C i C++. Oprogramowanie, które tworzymy od kilku lat, rozwinęło się w potężny pakiet narzędzi do numerycznego rozwiązywania równań różniczkowych cząstkowych na superkomputerach" [2]. Warto dodać, że PETSc na pewno będzie dalej rozwijane przez parę najbliższych lat, jako że autorom pakietu przyznano grant rządowy. Każdy użytkownik pakietu może zwrócić się do autorów o pomoc, ponadto ma do dyspozycji listę dyskusyjną oraz sporo dodatkowych wydawnictw, głownie w formie elektronicznej. Szczegóły można znaleźć na stronie WWW poświęconej PETSc w Argonne National Laboratory: http://www.mcs.anl.gov/petsc/
PETSc zawiera wiele składników podobnych do klas w C++. Każdy składnik dysponuje wydzieloną rodziną typów danych (na przykład wektorów) i operacji wykonywalnych na nich. Zarówno obiekty, jak operacje, opracowano na podstawie wieloletniego doświadczenia autorów (i użytkowników pakietu!) w rozwiązywaniu równań różniczkowych cząstkowych. Moduły PETSc operują obiektami takimi jak:
|
|
|
| 1. Generuj triangulację | |
| 2. Generuj macierze sztywności | Obiekty i operacje macierzowe (Mat) |
| 3. Generuj prawą stronę równania i przybliżenie początkowe | Obiekty i operacje wektorowe (Vec) |
| 4. Obejrzyj utworzone wektory i macierze | Przeglądarki obiektów PETSc (Viewer) |
| 5. Generuj preconditioner | Obiekty i operacje macierzowe i wektorowe |
| 6. Rozwiąż | Narzędzia do rozwiązywania równań liniowych i nieliniowych (SLES, SNES). Obiekty kontrolujące przebieg metod iteracyjnych (KSP, PC). Opcje wywołania i kontroli (Options). |
| 7. Obejrzyj wyniki |
W PETSc wszystkie funkcje nazywane są zgodnie z konwencją TypObiektuCzynność(), na przykład:
MatCreate() - generuje macierz
MatMult() - mnoży dwie macierze
VecAXBY() - wykonuje na wektorach
operację 
OptionsGetInt() - pobiera z opcji zmienną typu int
Nazwy są długie, co nie oszczędza nam stukania w klawiaturę, ale za to większość nazw funkcji jest zrozumiała i łatwa do zapamiętania, co wspomaga pisanie i czytanie programów korzystających z biblioteki PETSc.
Podręcznik składa się więc z dwóch części: w pierwszej wykładamy podstawy użytkowania PETSc i w oparciu o nie pokazujemy, jak można wykorzystać PETSc do rozwiązywania wielkich układów równań liniowych za pomocą nowoczesnych metod iteracyjnych. W drugiej części omawiamy bardziej zaawansowane techniki oraz metody wykorzystania PETSc do rozwiązywania układów równań nieliniowych. Obie części bogato ilustrujemy pouczającymi przykładami, związanymi najczęściej z eliptycznymi równaniami różniczkowymi cząstkowymi.
Wydaje się więc, że podręcznik ten trzeba czytać w sposób, hmm... monotoniczny, to znaczy "od początku do końca". Zachęcamy Czytelnika do pisania własnych programów i do testowania świeżo zdobytej wiedzy. W szczególności, dla dobrego wykonania ćwiczeń, którymi kończymy większość rozdziałów, warto jest zawsze zerknąć nie tylko do naszego podręcznika, ale też do man pages oraz oficjalnego manuala.
Nie da się ukryć, aby nauczyć się korzystać z PETSc, trzeba włożyć trochę wysiłku. Przede wszystkim, trzeba zacząć pisać programy. Dlatego zachęcamy do lektury poniższego pouczającego rozdziału.
program1.c
#include "sles.h"
static char help[]="Najprostszy program uzywajacy PETSc\n";
int main(int argc,char **args)
{
PetscInitialize(&argc,&args,PETSC_NULL,help);
PetscPrintf(MPI_COMM_WORLD,"PETSc działa i pozdrawia\n");
PetscFinalize();
}
hydra:/usr/home/students/bojko> program1 PETSc działa i pozdrawia hydra:/usr/home/students/bojko> _
Mimo, że kod źródłowy jest bardzo krótki, skompilowanie tego programu spowoduje wygenerowanie dość dużego pliku wynikowego, a to dlatego, że do każdego programu dołączany jest szereg procedur ułatwiających życie programistom i użytkownikom - chociażby zestaw procedur do śledzenia pracy programu. Na przykład, uruchomienie tego samego programu z opcją -help spowoduje wypisanie:
hydra: /usr/home/students/bojko> program1 -help -------------------------------------------------------------------------- PETSc Version 2.0.22, Released April 28, 1998. The PETSc Team: Satish Balay, Bill Gropp, Lois Curfman McInnes, Barry Smith Bug reports, questions: petsc-maint@mcs.anl.gov Web page: http://www.mcs.anl.gov/petsc/ See docs/copyright.html for copyright information See docs/changes.html for recent updates. See docs/troubleshooting.html hints for problems. See docs/manualpages/manualpages.html for help. Libraries linked from /usr/local/petsc/petsc-2.0.22/lib/libO/solaris ----------------------------------------------------------------------- Options for all PETSc programs: -on_error_abort: cause an abort when an error is detected. Useful only when run in the debugger -on_error_attach_debugger [dbx,xxgdb,ups,noxterm] start the debugger (gdb by default) in new xterm unless noxterm is given -start_in_debugger [dbx,xxgdb,ups,noxterm] start all processes in the debugger -debugger_nodes [n1,n2,..] Nodes to start in debugger -debugger_pause [m] : delay (in seconds) to attach debugger -display display: Location where graphics and debuggers are displayed -no_signal_handler: do not trap error signals -mpi_return_on_error: MPI returns error code, rather than abort on internal error -fp_trap: stop on floating point exceptions note on IBM RS6000 this slows run greatly -trdump: dump list of unfreed memory at conclusion -trmalloc: use our error checking malloc -trmalloc_off: don't use error checking malloc -trmalloc_nan: initialize memory locations with NaNs -trinfo: prints total memory usage -trdebug: enables extended checking for memory corruption -optionstable: dump list of options inputted -optionsleft: dump list of unused options -log[_all _summary]: logging objects and events -log_summary_exclude: <vec,mat,sles,snes> -log_trace [filename]: prints trace of all PETSc calls -log_info: print informative messages about the calculations -v: prints PETSc version number and release date -options_file <file>: reads options from file ----------------------------------------------- Najprostszy program uzywajacy PETSc PETSc działa i pozdrawia hydra:/usr/home/students/bojko>_
Zanim rozpoczniemy nasze bliskie spotkanie z PETSc, jeszcze krótko omówimy sposób kompilowania i uruchamiania programów korzystających z biblioteki PETSc. Po pierwsze aby móc skompilować swój program należy ustawić zmienne środowiskowe PETSC_DIR, PETSC_ARCH, BLAS_DIR, LAPACK_DIR jak następuje:
setenv PETSC_DIR /usr/local/petsc/petsc-2.0.22 setenv PETSC_ARCH solaris setenv BLAS_DIR /usr/local/petsc/blaslapack setenv LAPACK_DIR /usr/local/petsc/blaslapack
makefile BOPT=O include $(PETSC_DIR)/bmake/$(PETSC_ARCH)/base CFLAG = $(CPPFLAGS) -D__SDIR__='"$(LOCDIR)"' $(BS_INCLUDE) LIBBASE = libpetscsles LOCDIR = /usr/home/students/bojko/source/ program1: program1.o -$(CLINKER) -o program1 program1.o $(PETSC_LIB) $(RM) program1.oOczywiście, parametr LOCDIR powinien być ustawiony na ścieżkę z Twoimi plikami źródłowymi.
 .
Pozwoli to na uruchomienie prostego programu rozwiązującego układ równań
powstałych z dyskretyzacji zagadnienia Poissona
.
Pozwoli to na uruchomienie prostego programu rozwiązującego układ równań
powstałych z dyskretyzacji zagadnienia Poissona
metodą różnic skończonych.
Następnie omówione zostaną szczegółowo struktury PETSc pozwalające na wybór metod iteracyjnych i włączanie w nie preconditionerów (także własnych). Ostatni rozdział poświęcony zostanie na szczegółowy opis istniejącego w pakiecie preconditionera addytywnej metody Schwarza, wykorzystującego metodę dekompozycji obszaru.
... Vec X; ...
Najprostszą funkcją służącą do wygenerowania wektora jest funkcja:
Pierwszy argument to informacja dla MPI (Message Passing Interface) o sposobie komunikacji między procesami. Nie warto zagłębiać się w subtelności z tym związane. Ważne jest, że z argumentem MPI_COMM_WORLD funkcja działa poprawnie zarówno na maszynach wieloprocesorowych, jak i jednoprocesorowych i we wszystkich podobnych przypadkach rozważanych w tym podręczniku będzie używana ta właśnie wartość. Drugi argument, n, jest liczbą elementów wektora przechowywanych w procesorze, który wywołał funkcję; możemy podać wartość PETSC_DECIDE, gdy chcemy aby pakiet ustawił tę wielkość automatycznie. PETSC_DECIDE jest idealną wartością dla n, gdy program nasz ma działać przede wszystkim w wersji sekwencyjnej. Trzeci argument, N, jest liczbą współrzędnych całego wektora (można wstawić PETSC_DETERMINE, wtedy zostanie ona określona na podstawie liczby procesorów i wartości n). Ostatnim argumentem jest wskaźnik do zmiennej typu wektor. Wektor będzie składał się ze współrzędnych typu Scalar.
... VecCreate(MPI_COMM_WORLD,PETSC_DECIDE,100,&X); ...
... double x[100]; ...a drugi sposób to dynamicznie:
... double *x; ... x=(double*)calloc(100,sizeof(double)); ...Sposób deklarowania wektorów (i nie tylko) w PETSc jest podobny do tej drugiej metody. Faktyczne przydzielenie pamięci następuje w momencie wywołania funkcji VecCreate().
Funkcja VecCreate() jest zaprojektowana tak, aby automatycznie rozproszyć wektor pomiędzy dostępne procesory (procesy). Pakiet sam zadecyduje czy wygenerować wektor sekwencyjny, czy równoległy, a jeśli równoległy, to ile elementów wektora będzie przechowywanych w poszczególnych procesorach.
Po wygenerowaniu wektora można przypisać wartości poszczególnym współrzędnym. Służą do tego dwie funkcje: VecSet(Scalar *val, Vec X) która nada wszystkim współrzędnym jednakową wartość val lub funkcja VecSetValue(Vec X, int idx, Scalar val, InsertMode mode). Pierwszy argument tej funkcji to wektor w który wstawiamy wartość, drugi to numer współrzędnej, trzeci wartość na tej współrzędnej. Czwarty argument to tryb wstawiania wartości, Możliwe są dwie wartości: ADD_VALUES - dodaj wartość do istniejącej, INSERT_VALUES - wstaw nową wartość.
 ,
w który zostaną wstawione wartości
,
w który zostaną wstawione wartości

a na końcu wektor zostanie wypisany na konsolę.
#include "sles.h"
static char help[]="przyklad2\n";
int main(int argc,char **args)
{
Vec x;
Scalar val;
int i,n=10;
/* inicjalizacja */
PetscInitialize(&argc,&args,PETSC_NULL,help);
/* wygenerowanie wektora */
VecCreate(MPI_COMM_WORLD,PETSC_DECIDE,n,&x);
/* przypisanie wartosci */
for(i=0;i<n;i++)
{
if(i>0)val=(Scalar)1/i; else val=1;
VecSetValue(x,i,val,INSERT_VALUES);
}
/* wypisuje wektor na konsolę */
VecView(x,VIEWER_STDOUT_SELF);
/* zakończenie */
VecDestroy(x);
PetscFinalize();
return(0);
}
To dostaniemy po uruchomieniu przykładu:
hydra:/usr/home/bin/> przykład 1 1 0.5 0.333333 0.25 0.2 0.166667 0.142857 0.125 0.111111 hydra:/usr/home/bin/> _
Po zadeklarowaniu, wygenerowaniu i przypisaniu wartości wektorowi x, na zakończenie programu wypisujemy go na ekran przy użyciu przeglądarki do wektorów VecView().
Aby wygenerować kilka takich samych wektorów (na przykład wektor rozwiązania równania i wektor prawej strony - oba są tego samego rozmiaru) można użyć funkcji VecDuplicate(Vec stary,Vec *nowy) lub VecDuplicateVecs(Vec stary,int n, Vec **nowe) pierwsza z tych funkcji utworzy wektor nowy o takiej samej strukturze jak wektor stary, to znaczy o tym samym rozmiarze i tak samo rozproszony pomiędzy procesorami. Druga utworzy n nowych wektorów o takiej samej strukturze jak stary i zapisuje w tablicy wektorów nowe. Powielenia struktury wektora nie należy mylić z kopiowaniem jego wartości. Do tego służy funkcja VecCopy(Vec x,Vec y), kopiująca wartości z wektora x do y. Wektory x i y musza w tym wypadku mieć taką samą strukturę, co znaczy, że uprzednio muszą być utworzone, na przykład przy pomocy funkcji VecCreate() lub VecDuplicate().
Nie istnieje, niestety, funkcja VecGetValues(), która pozwalałaby na pobranie wartości z wektora. W przypadku równoległym trzeba stosować specjalne funkcje zbierające i rozsyłające dane po procesorach. W przypadku sekwencyjnym można używać funkcji VecGetArray(Vec x,Scalar **val), która zwraca wskaźnik do tablicy elementów wektora przechowywanych w procesorze który wywołał tę procedurę. Po zakończeniu wszelkich operacji na tablicy elementów trzeba ją z powrotem wstawić do wektora funkcją VecRestoreArray(Vec x, Scalar **val).
Przydatna jest też funkcja VecView(Vec v, Viewer viewer), która pokazuje wektor v przy pomocy przeglądarki viewer. Istnieją trzy predefiniowane przeglądarki:
|
|
|
| VIEWER_STDOUT_SELF | standardowa tekstowa; wypisuje wektor na konsolę |
| VIEWER_STDOUT_WORLD | standardowa tekstowa zsynchronizowana, przydatna w przypadku programów równoległych |
| VIEWER_DRAWX_WORLD | graficzna; rysuje wektor w oknie graficznym jako funkcję współrzędnej |
Gdy wektor nie jest już potrzebny, należy zwolnić pamięć którą zajmuje funkcją VecDestroy(Vec x) lub - w przypadku tablicy wektorów - VecDestroyVecs(Vec *wektory,int n).
|
|
|
| VecAXPY(Scalar *a,Vec x,Vec y); |
 |
| VecAYPX(Scalar *a,Vec x,Vec y); |
 |
| VecWAXPY(Scalar *a,Vec x,Vec y,Vec w); |
 |
| VecScale(Scalar *a,Vec x); |
 |
| VecDot(Vec x,Vec y,Scalar *r); |
 |
| VecTDot(Vec x,Vec y,Scalar *r); |
 |
| VecNorm(Vec x,NormType norm,Double *r);
norm |
 |
| VecSum(Vec x, Scalar *r); |
 |
| VecCopy(Vec x, Vec y); |
 |
| VecSwap(Vec x, Vec y); |
 |
| VecPointwiseMult(Vec x, Vec y, Vec w); |
 |
| VecPointwiseDivide(Vec x, Vec y, Vec w); |
 |
| VecMax(Vec x, int *idx, double *r); |
 |
| VecMin(Vec x, int *idx, double *r); |
 |
| VecAbs(Vec x); |
 |
| VecReciprocal(Vec x); |
 |
| VecShift(Scalar *s, Vec x); |
 |
static char help[]="przyklad 4.6";
#include "sles.h"
#include <stdio.h>
int main(int argc,char **args)
{
Vec x,y;
Scalar val,*wartosci;
int i,n=5,*indeksy,flg;
PetscInitialize(&argc,&args,PETSC_NULL,help);
VecCreate(MPI_COMM_WORLD,PETSC_DECIDE,n,&x);
VecDuplicate(x,&y);
indeksy=(int*)calloc(n/2,sizeof(int));
wartosci=(Scalar*)calloc(n/2,sizeof(Scalar));
val=0.5;
VecSet(&val,x);
VecView(x,VIEWER_STDOUT_SELF);
PetscPrintf(MPI_COMM_WORLD,"\n");
VecCopy(x,y);
/* przygotowanie danych */
for(i=0;i<n/2;i++)
{
if(i>0)wartosci[i]=(Scalar)1/i;
else wartosci[i]=1;
indeksy[i]=2*i;
}
/* Wstawienie danych do wektora */
VecSetValues(y,n/2,indeksy,wartosci,ADD_VALUES);
VecAssemblyBegin(y);
VecAssemblyEnd(y);
VecView(y,VIEWER_STDOUT_SELF);
PetscPrintf(MPI_COMM_WORLD,"\n");
/* policzenie różnicy dwóch wektorów */
val=-1;
VecAXPY(&val,x,y);
/*wypisanie wektorów */
VecView(y, VIEWER_STDOUT_SELF);
PetscPrintf(MPI_COMM_WORLD,"\n");
/*zakończenie*/
VecDestroy(x);
VecDestroy(y);
free(indeksy);
free(wartosci);
PetscFinalize();
return(0);
}
hydra:/usr/home/> przyklad 0.5 0.5 0.5 0.5 0.5 1.5 0.5 1.5 0.5 0.5 1 0 1 0 0 hydra:/usr/home/> _Ten program tworzy dwa wektory x,y o długości 5 (jakie są ich współrzędne?). Następnie, kładziemy
 , a
następnie wektory są wypisywane na konsolę. Zwróćmy uwagę, w jaki sposób
do odejmowania wektorów używamy funkcji VecAXPY(): realizując operację
, a
następnie wektory są wypisywane na konsolę. Zwróćmy uwagę, w jaki sposób
do odejmowania wektorów używamy funkcji VecAXPY(): realizując operację !
!
 ,
Obejrzyj te wektory na konsoli i w okienku graficznym. (Uwaga: w pliku
<math.h> jest zdefiniowana stała M_PI zawierająca
przybliżenie
,
Obejrzyj te wektory na konsoli i w okienku graficznym. (Uwaga: w pliku
<math.h> jest zdefiniowana stała M_PI zawierająca
przybliżenie  )
) .
(Zauważ, że taki wektor można potraktować jako dyskretyzację funkcji
.
(Zauważ, że taki wektor można potraktować jako dyskretyzację funkcji  na
odcinku [0,1]). Obejrzyj ten wektor na konsoli i w okienku graficznym.
Eksperymentuj dla różnych wartości N.
na
odcinku [0,1]). Obejrzyj ten wektor na konsoli i w okienku graficznym.
Eksperymentuj dla różnych wartości N. .
Przez analogię z poprzednim można ten wektor traktować jako dyskretyzację
funkcji
.
Przez analogię z poprzednim można ten wektor traktować jako dyskretyzację
funkcji  na kwadracie [0,1]2
z siatką o kroku h = 1/(N-1). Obejrzyj y w okienku graficznym.
Zobacz jak trudno zgadnąć, jaką funkcję dyskretyzuje y, stąd wniosek,
że do takiej wizualizacji trzeba używać innych narzędzi. Dowiedz się, jakie
programy wizualizacyjne są dostępne na Twoim komputerze i jak z nich korzystać.
na kwadracie [0,1]2
z siatką o kroku h = 1/(N-1). Obejrzyj y w okienku graficznym.
Zobacz jak trudno zgadnąć, jaką funkcję dyskretyzuje y, stąd wniosek,
że do takiej wizualizacji trzeba używać innych narzędzi. Dowiedz się, jakie
programy wizualizacyjne są dostępne na Twoim komputerze i jak z nich korzystać. , b)
, b) ,
c)
,
c) wektorów x i y
z poprzednich ćwiczeń. Jakich relacji między tymi normami można się spodziewać?
wektorów x i y
z poprzednich ćwiczeń. Jakich relacji między tymi normami można się spodziewać?Po zadeklarowaniu zmiennej jako macierz:
... Mat A; ...
 ,
gdzie m to liczba wierszy, a n kolumn. Wstawianie wartości
do macierzy odbywa się analogicznie jak w przypadku wektorów, przy pomocy
funkcji MatSetValue(Mat A,int row,int col,Scalar val,InsertMode
mode). Ta funkcja wpisuje w macierz A wartość val
w miejsce row,col. Ostatni argument określa, podobnie jak
w przypadku wektorów, sposób wstawiania wartości do macierzy i może przyjąć
wartości INSERT_VALUES lub ADD_VALUES. Do zwolnienia
pamięci zajmowanej przez macierz służy funkcja MatDestroy(Mat A),
która działa tak samo jak jej odpowiednik dla wektorów.
,
gdzie m to liczba wierszy, a n kolumn. Wstawianie wartości
do macierzy odbywa się analogicznie jak w przypadku wektorów, przy pomocy
funkcji MatSetValue(Mat A,int row,int col,Scalar val,InsertMode
mode). Ta funkcja wpisuje w macierz A wartość val
w miejsce row,col. Ostatni argument określa, podobnie jak
w przypadku wektorów, sposób wstawiania wartości do macierzy i może przyjąć
wartości INSERT_VALUES lub ADD_VALUES. Do zwolnienia
pamięci zajmowanej przez macierz służy funkcja MatDestroy(Mat A),
która działa tak samo jak jej odpowiednik dla wektorów.
#include "sles.h"
static char help[]="przykład_3\n";
int main(int argc,char **args)
{
Mat A;
int i,n=5;
/* inicjalizacja */
PetscInitialize(&argc,&args,PETSC_NULL,help);
/* wygenerowanie macierzy trójdiagonalnej*/
MatCreate(MPI_COMM_WORLD,n,n,&A);
/* wypełniamy macierz wierszami */
for(i=0;i<n;i++)
{
MatSetValue(A,i,i,2,INSERT_VALUES);
if(i<n-1)MatSetValue(A,i,i+1,1,INSERT_VALUES);
if(i>0)MatSetValue(A,i,i-1,1,INSERT_VALUES);
}
MatAssemblyBegin(A,MAT_FINAL_ASSEMBLY);
MatAssemblyEnd(A,MAT_FINAL_ASSEMBLY);
/* wypisuje macierz na konsolę */
MatView(A,VIEWER_STDOUT_SELF);
/* zakończenie */
MatDestroy(A);
PetscFinalize();
return(0);
}
hydra:/usr/home/bin/> przykład row 0: 0 2 1 -1 row 1: 0 -1 1 2 2 -1 row 2: 1 -1 2 2 3 -1 row 3: 2 -1 3 2 4 -1 row 4: 3 1 4 2 hydra:/usr/home/bin/> _
 Rysunek
1. Działanie funkcji MatSetValues().
Rysunek
1. Działanie funkcji MatSetValues().Przyjrzyjmy się, jak działa przeglądarka macierzy MatView() w trybie tekstowym: macierz jest wypisywana wierszami. W każdym wierszu podawane są tylko niezerowe elementy (bardzo słusznie!); dlatego format wydruku jest następujący:
... row wiersz: kolumna wartość kolumna wartość kolumna wartość ....
Ta funkcja rozprasza w macierzy A macierz o rozmiarze mxn zapisaną w tablicy val w miejsca zapisane w tablicach row i col. Sposób działania tej funkcji wyjaśnia rysunek 1.
Aby wartości zostały fizycznie wpisane do macierzy należy, zawsze wywołać funkcje: MatAssemblyBegin(Mat A,MatAssemblyType type) i MatAssemblyEnd(Mat A, MatAssemblyType type). Zmienna type może przyjąć dwie wartości: MAT_FLUSH_ASSEMBLY bądź MAT_FINAL_ASSEMBLY. Pierwsza wartość jest używana, gdy zmieniamy tryb wstawiania wartości (INSERT_VALUES na ADD_VALUES, bądź na odwrót), druga - gdy kończymy wstawianie wartości i macierz ma zostać użyta.
 Rysunek
2. Macierz trójdiagonalna z poprzedniego przykładu w przeglądarce graficznej.
Rysunek
2. Macierz trójdiagonalna z poprzedniego przykładu w przeglądarce graficznej.Funkcja MatCreate() tworzy szkielet macierzy rozrzedzonej pewnego (domyślnego) rodzaju. Użytkownik może też samemu wpłynąć na sposób generowania macierzy. Na przykład, można zaznaczyć jak duże kawałki macierzy mają być przechowywane w poszczególnych procesach, czy też, jak macierze mają być składowane: wierszami czy kolumnami. Szczegółowy opis można znaleźć w manualu [2] i man pages [3]. Dla potrzeb początkującego użytkownika przydatna jest jeszcze funkcja MatCompress(Mat A), która poddaje macierz kompresji, tak aby zajmowała jak najmniej miejsca w pamięci, gdyż funkcja MatCreate alokuje 10 razy ilość wierszy macierzy, co z reguły jest za dużo. Warto zatem po zakończeniu wpisywania wartości i wywołaniu funkcji MatAssemblyEnd(), wywołać funkcję MatCompress(). W przypadku gdy wiemy, że macierz zajmie więcej miejsca niż domyślne "10 razy ilość wierszy" trzeba użyć funkcji MatCreateSeqAIJ(), lub podobnej, ponieważ alokowanie dodatkowego miejsca w czasie wstawiania wartości do macierzy może spowodować nawet 50 krotne (!) spowolnienie działania funkcji.
Jak już wspomnieliśmy, dla macierzy istnieje funkcja MatView(Mat A,Viewer view), która pokazuje macierz w przeglądarce. Przeglądarka VIEWER_DRAWX_WORLD pokazuje, w okienku graficznym, strukturę niezerowych miejsc macierzy. Rysunek 2 pokazuje efekt przeglądania w trybie graficznym macierzy z poprzedniego przykładu.
|
|
|
| MatEqual(Mat A,Mat B) | sprawdza czy A=B |
| MatMult(Mat A, Vec x, Vec y) |
 |
| MatMultAdd(Mat A, Vec x,Vec y, Vec z) |
 |
| MatMultTrans(Mat A, Vec x,Vec y) |
 |
| MatMultTransAdd(Mat A, Vec x,Vec y, Vec z) |
 |

po czym mnożony jest przez nią wektor złożony z samych jedynek, a efekt zostaje wypisany na konsolę.
#include "sles.h"
int main(int argc,char **args)
{
Vec x,y;
Mat A;
int i,k,l,n=4,m=4,flg,col[5];
Scalar val[5];
/* inicjalizacja */
PetscInitialize(&argc,&args,PETSC_NULL,PETSC_NULL);
/* wektory przydatne przy demonstracji */
VecCreate(MPI_COMM_WORLD,PETSC_DECIDE,n*m,&x);
VecDuplicate(x,&y);
val[0]=1.0;
VecSet(val,x);
/* Tworzenie macierzy laplasjanu na prostokacie mxn */
MatCreate(MPI_COMM_WORLD,n*m,n*m,&A);
val[0] = -1.0; val[1] = -1.0; val[2] = 4.0;
val[3]=-1.0;val[4]=-1.0;
for (i=0; i<n*m; i++ )
{
col[0]=i+1; col[1]=i-1; col[2]=i;
col[3]=i+m; col[4]=i-m;
l=0;k=5;
if(i<m)k=4;
if((i+1)>(n-1)*m){k=4;col[3]=i-m;}
if((i%m)==0){l=1;k=k-1;col[1]=i+1;}
if((i+1)%m==0)if(i!=0){l=1;k=k-1;}
MatSetValues(A,1,&i,k,&col[l],&val[l],INSERT_VALUES);
}
/* Składanie macierzy*/
MatAssemblyBegin(A,MAT_FINAL_ASSEMBLY);
MatAssemblyEnd(A,MAT_FINAL_ASSEMBLY);
/*Mnożenie wektora x przez macierz A (y=A*x)*/
MatMult(A,x,y);
/*Wypisanie wektora y na konsolę*/
VecView(y,VIEWER_STDOUT_SELF);
/* zakończenie */
VecDestroy(x);
MatDestroy(A);
PetscFinalize();
return(0);
}
hydra:/usr/home/>p5.6 2 1 1 2 1 0 0 1 1 0 0 1 2 1 1 2 hydra:/usr/home/> _

jest macierzową reprezentacją (w standardowej bazie R3) następującego operatora:

Taka definicja jest szczególnie ważna w przypadku macierzy rozrzedzonych, w których powtarzają się elementy, gdyż dzięki temu nie musimy tracić miejsca w pamięci na powtarzające się wartości. Na przykład macierz powstała z dyskretyzacji jednowymiarowego laplasjanu ma (z dokładnością do stałej) postać:

Działanie tą macierzą na wektor można zapisać następująco:
 ,
,gdzie

Autorzy pakietu przewidzieli możliwość definiowania macierzy w ten sposób. Użytkownik informuje pakiet jedynie o rozmiarze macierzy i podaje poszczególne funkcje realizujące operacje na tej macierzy (czy tą macierzą). Istotne jest to, że taka macierz jest traktowana tak samo jak inne rodzaje macierzy. Dopóki program nie usiłuje wykonać na nich operacji niewykonalnej, to znaczy nie zdefiniowanej przez użytkownika, to nie ma absolutnie żadnej różnicy w posługiwaniu się tą macierzą i macierzą "zwykłą". Macierze definiowane w duchu operatorowym zwane są powłokowymi (ang. shell matrix).
Mając zadeklarowaną zmienną typu Mat,
możemy utworzyć ją w postaci macierzy powłokowej. Do tego służy funkcja:
MatShellCreate(MPI_Comm com,int m,int n,int M,int N,void *ctx,Mat
*mat)
Pierwszy argument to kanał komunikacyjny MPI (np. MPI_COMM_WORLD), drugi i trzeci argument (m i n) to rozmiar lokalny macierzy, to znaczy rozmiar części macierzy zapisanej w procesorze wywołującym tę funkcję. Czwarty i piąty argument (M i N) to globalny rozmiar macierzy. Oczywiście, w wersji sekwencyjnej będziemy mieli n=N i m=M; ponadto (zazwyczaj) nasze macierze będą kwadratowe więc wtedy dodatkowo m=n=M=N. Szósty argument (ctx) to wskaźnik do kontekstu związanego z macierzą, lub PETSC_NULL, gdy nie korzystamy z kontekstu. Ostatni argument to wskaźnik do zmiennej typu Mat z którą będzie powiązana macierz powłokowa.
Do definiowania operacji, którą można wykonać na macierzy powłokowej służy funkcja:
MatShellSetOperation(Mat mat,MatOperation op, void *f)
Pierwszy argument tej funkcji to zmienna powiązana z obiektem typu Mat, dla którego definiujemy operację. Drugi argument to nazwa operacji, a trzeci to wskaźnik do funkcji wykonującej tą operację. Zdefiniować można 63 operacje, w poniższej tabeli podanych jest kilka ważniejszych:
|
|
|
MATOP_MULT |
Pomnożenie wektora przez macierz |
MATOP_EQUAL |
Sprawdzenie czy dwie macierze są sobie równe |
MATOP_GET_SIZE |
Zwrócenie rozmiaru macierzy |
MATOP_DESTROY |
Zwolnienie pamięci zajmowanej przez macierz |
MATOP_VIEW |
Pokazanie macierzy |
MATOP_NORM |
Norma macierzy |
MATOP_GET_DIAGONAL |
Zwrócenie diagonali macierzy |
MATOP_DIAGONAL_SCALE |
Pomnożenie macierzy przez macierz diagonalną |
MATOP_SET_VALUES |
Wstawienie wartości do macierzy |
#include "sles.h"
#define N=100
/****************************************************
Procedura mnozenia przez macierz
****************************************************/
int mult(Mat A,Vec x,Vec y)
{
VecCopy(x,y);
return(0);
}
/****************************************************
Program glowny
****************************************************/
main(int argc,char **args)
{
Mat A;
Vec v,b;
PetscInitialize(&argc,&args,PETSC_NULL,PETSC_NULL);
/* ...pominiete linie kodu: tworzenie wektora b i przypisanie mu
wartosci... */
/* tworzenie macierzy powłokowej */
MatCreateShell(MPI_COMM_WORLD,N,N,N,N,PETSC_NULL,&A);
MatShellSetOperation(A,MATOP_MULT,mult);
/* zastosowanie */
MatMult(A,v,b);
PetscFinalize();
}

#include "sles.h"
#include <stdio.h>
/****************************************************
Procedura mnozenia przez macierz
****************************************************/
int mult(Mat A,Vec x,Vec y)
{
int i,N,*idx;
Scalar *wx,*wy;
VecGetSize(x,&N);
VecGetArray(x,&wx);
VecGetArray(y,&wy);
for(i=0;i<N;i++)
{
wy[i] = 2*wx[i];
if(i>0) wy[i] -= wx[i-1];
if(i<N) wy[i] -= wx[i+1];
}
VecRestoreArray(x,&wx);
VecRestoreArray(y,&wy);
}
/****************************************************
Program glowny
****************************************************/
int main(int argc,char **args)
{
Mat A;
int N=10;
Scalar val;
Vec x,y;
PetscInitialize(&argc,&args,PETSC_NULL,PETSC_NULL);
/* tworzenie macierzy powłokowej */
MatCreateShell(MPI_COMM_WORLD,N,N,N,N,0,&A);
MatShellSetOperation(A,MATOP_MULT,mult);
/* Wektory przydatne przy prezentacji */
VecCreate(MPI_COMM_WORLD,PETSC_DECIDE,N,&x);
VecDuplicate(x,&y);
val=1;
VecSet(&val,x);
/* uzycie macierzy powlokowej */
MatMult(A,x,y);
VecView(y,VIEWER_STDOUT_SELF);
/* Zakończenie */
VecDestroy(x); VecDestroy(y);
MatDestroy(A);
PetscFinalize();
return(0);
}
To dostaniemy po uruchomieniu przykładu:
hydra:/usr/home/> przykład 1 0 0 0 0 0 0 0 0 1 hydra:/usr/home/> _


obejrzyj ją w okienku, a następnie na konsoli. Naucz się odczytywać te widoki.
 ,
gdzie
,
gdzie  . Napisz procedurę
GenLap1D(Mat A, int h) która generuje macierz NxN dyskretyzacji
1-wymiarowego Laplasjanu.
. Napisz procedurę
GenLap1D(Mat A, int h) która generuje macierz NxN dyskretyzacji
1-wymiarowego Laplasjanu.
metodą różnic skończonych. Wskazówka: użyj funkcji GenLap1D() a następnie dodaj wartości pochodzące z dyskretyzacji pierwszej pochodnej.

Użyj obu macierzy do dyskretyzacji operatora L z ćwiczenia E) w wersji

(Najwygodniej zdefiniować go jako macierz powłokową!)
Aby go rozwiązać, musimy utworzyć wektory rozwiązania i prawej strony oraz macierz zadania, a następnie spiąć te wszystkie dane ze sobą i zlecić pakietowi rozwiązanie tego układu. Do tego służą właśnie obiekty typu SLES.
Aby utworzyć obiekt typu SLES trzeba, podobnie jak w poprzednich przypadkach, najpierw zadeklarować odpowiednią zmienną:
... SLES MojSles; ...
Pierwszy argument funkcji SLESCreate() to, podobnie jak w przypadku innych obiektów, kanał komunikatora MPI, u nas zawsze będzie to MPI_COMM_WORLD. Drugi to wskaźnik do uprzednio zadeklarowanej zmiennej typu SLES.
Następną czynnością, jaką należy wykonać, jest przekazanie macierzy zadania do SLESu. Robi się to przy pomocy funkcji:
SLESSetOperators(SLES MojSles,Mat A,Mat P, MatStructure flag);
|
|
|
| SAME_PRECONDITIONER | Macierz P jest cały czas taka sama w czasie procesu iteracyjnego. Ta opcja jest dla tych, którzy używają różnych macierzy A i P. Opcja ta np. oszczędza pracę przy niekompletnym rozkładzie Choleskiego stosowanym jako preconditioner: rozkład jest robiony tylko raz. |
| SAME_NONZERO_PATTERN | w trakcie iteracji macierz P ma taką samą strukturę miejsc niezerowych |
| DIFFERENT_NONZERO_PATTERN | w trakcie iteracji macierz P zmienia strukturę |
Powyższe przygotowania wystarczają, aby SLES rozwiązał układ równań z macierzą A i prawą stroną b. W tym celu należy wywołać funkcję SLESSolve(SLES MojSles,Vec b,Vec x,int *its). Wynik zostanie zapisany w wektorze x.
W zmiennej its zostanie zapisana liczba wykonanych iteracji. Po zakończeniu obliczeń należy zwolnić pamięć zajmowaną przez SLES funkcją: SLESDestroy(SLES MojSles), gdzie argumentem jest (niepotrzebny już) obiekt MojSles. Ważne jest, że jeden SLES może służyć do rozwiązania wielu układów równań, na przykład kilku układów z tą samą macierzą i inną prawą stroną.
#include "sles.h"
int main(int argc,char **args)
{
Vec x,b;
Mat A; /* macierz układu równań */
SLES MojSles; /* kontekst SLESu */
int its;
Scalar val;
PetscInitialize(&argc,&args,PETSC_NULL,PETSC_NULL);
/* Tworzenie kontekstu SLES */
SLESCreate(MPI_COMM_WORLD,&MojSles);
/* tutaj trzeba utworzyć wektory (str. 12)
i macierze, (str. 17) */
/*Ustawienie operatorów */
SLESSetOperators(MojSles,A,A,DIFFERENT_NONZERO_PATTERN);
SLESSetFromOptions(MojSles);
/* rozwiązanie układu liniowego */
SLESSolve(MojSles,b,x,&its);
/*Tu z kolei należy zwolnić pamięć*/
SLESDestroy(MojSles);
PetscFinalize();
return(0);
}

na siatce o kroku h. Testuj błąd rozwiązania
w normie max i l2 dla f odpowiadającym
rozwiązaniu dokładnemu
a) sin(x),
b) x2+y2.
Sprawdź, jak zmienia się liczba iteracji w zależnościod h. Obejrzyj
wektory rozwiązania i błędu w okienku graficznym.

Eksperymentuj z różnymi sposobami implementacji macierzy (klasycznie i powłokowo).
hydra:/usr/home/bin> make moj_program
Aby jednak to działało, należy jednak napisać własny plik tekstowy nazwany Makefile w którym podane zostaną (ale tylko raz) wszystkie parametry potrzebne do kompilowania i linkowania programu. Przykładowy plik Makefile, pozwalający na skompilowanie programu z dołączeniem bibliotek PETSc podajemy poniżej. Zmienna BOPT mówi jaka wersja programu ma być tworzona. BOPT=O oznacza program zoptymalizowany, BOPT=g oznacza wersję do odpluskwiania (debuggingu). Ważne jest, żeby gotowy (tzn. odpluskwiony) program kompilować z opcją BOPT=O, gdyż to daje kilkukrotnie mniejszy (około 5 razy) i szybszy kod wynikowy. Oczywiście, aby w ogóle dało się skompilować program, muszą być ustawione zmienne środowiskowe opisane w rozdziale "Zaczynamy" na stronie 7. Tworzenie Makefile'ów jest odrębną sztuką. Zamiast ją zgłebiać, proponujemy pisanie własnych przez prostą modyfikację poniższego:
BOPT=O include $(PETSC_DIR)/bmake/$(PETSC_ARCH)/base CFLAG = $(CPPFLAGS) D__SDIR__='"$(LOCDIR)"' $(BS_INCLUDE) LIBBASE = libpetscsles LOCDIR = /usr/home/students/bojko/source/ moj_program: moj_program.o $(CLINKER) -o moj_program moj_program.o $(PETSC_LIB) $(RM) moj_program.oBardziej rozbudowane Makefile, umożliwiające kompilację kilku różnych programów, znajdują się na stronach 41 i 55.
hydra:/usr/home/bin> moj_program [opcje]
Drugi sposób stosujemy, gdy uruchamiamy program na maszynie wieloprocesorowej z zainstalowaną biblioteką MPI. Wówczas program uruchamia się komendą:
hydra:/usr/home/bin> mpirun -np 4 moj_program [opcje]
Opcja -np 4 oznacza liczbę procesorów, na których będzie uruchomiony moj_program (w tym wypadku 4). Dalej podajemy nazwę programu i ewentualne opcje samego programu.
#include "sles.h"
Jak już wspomnieliśmy, dzięki użyciu funkcji main() z argumentami argc, args oraz wywołaniu funkcji PetscInitialize(), tekst z linii komend zostanie przekazany bezpośrednio do programu i potraktowany jako opcje, co pozwala na sterowanie wieloma parametrami programu. Można zmieniać rozmiar zadania, podawać dodatkowe parametry itp.
W pełnym wykorzystaniu opcji pomocne będą funkcje: OptionsGetXXX(char* pre, char *name, XXX *zmienna,int *flag) gdzie w miejscu XXX wstawiamy typ wartości podawanej z linii komend: Int, Double, Scalar, String, DoubleArray. Na przykład funkcja
.... OptionsGetInt(PETSC_NULL,"-N",&rozmiar); ....
Argumenty funkcji OptionsGetXXX() mają następujące znaczenie: pierwszy to przedrostek, u nas będzie zawsze ustawiony na PETSC_NULL. Drugi to poszukiwany ciąg znaków, a trzeci argument to wskaźnik zmiennej do której będzie przekazywany wynik. Ostatni argument to wskaźnik do zmiennej, w której zapisane zostanie czy przypisanie wartości miało miejsce, czy nie. Przydatna jest też funkcja OptionsHasName(char* pre, char *name,int *flag), która sprawdza, czy dany ciąg znaków name pojawił się w linii komend i wynik zapisuje w zmiennej flag.
Ważnym ułatwieniem w pisaniu programów jest to, że wszystkie funkcje w PETSc zwracają kody błędów. Jest też makro CHKERRA(int ierr), która w przypadku wystąpienia błędu poda numer linii programu i nazwę pliku z kodem źródłowym oraz krótki opis błędu. Jeżeli zatem każdą funkcję PETSc wywołujemy następująco:
... ierr = VecCreate(MPI_COMM_WORLD,PETSC_DECIDE,N,&v); CHKERRA(ierr); ierr = VecSet(&val,v); CHKERRA(ierr); ...
Opcja -sles_view spowoduje wypisanie na konsolę informacji na temat SLESu
Opcja -vec_view po wywołaniu w programie funkcji VecAssemblyBegin(v) zostanie wypisany na konsolę wektor v. Podobna do niej -vec_view_draw narysuje w oknie graficznym wektor jako funkcję indeksu.
Opcja -mat_view po wywołaniu w programie funkcji MatAssemblyBegin(A,FINAL_ASSEMBLY) zostanie wypisana na konsolę macierz A. Podobna do niej -mat_view_draw narysuje w oknie graficznym strukturę miejsc niezerowych macierzy.
Opcja -draw_pause <n> spowoduje zatrzymanie przeglądarki graficznej (zarówno wywoływanej opcją z linii komend jak i z programu) na n sekund. Jeżeli n=0 to przeglądarka czeka na kliknięcie klawiszem myszki. To bardzo przydatna opcja, gdyż często przeglądarka jest zamykana zaraz po zakończeniu rysowania, co w wielu przypadkach nie wystarcza nawet na zauważenie, że okno pojawiło się na ekranie.
Opcją -ksp_type <typ> możemy zmienić rodzaj metody iteracyjnej używanej do rozwiązywania układu równań (szczegóły w rozdziale 0 na stronie 29) Inne opcje będziemy omawiać na bieżąco.
Opis opcji dostępnych w każdym z programów jest wypisywany gdy uruchamiamy program z opcją help.
#include "sles.h"
static char help[]="Pomoc";
int main(int argc,char **args)
{
int N=5,pc_flag=0;
double af=0.1;
PetscInitialize(&argc,&args,PETSC_NULL,help);
OptionsGetInt(PETSC_NULL,"-N",&N);
OptionsGetDouble(PETSC_NULL,"-alfa",&af);
OptionsHasName(PETSC_NULL,"-bez_pc",&pc_flag);
PetscPrintf(MPI_COMM_WORLD,"rozmiar=%i,af=%d,pc_flag=%i\n",N,af,pc_flag);
PetscFinalize();
return(0)
}
hydra:/usr/home/bin/> program -help -N 10 -alfa 0.22
spowoduje przypisanie zmiennej rozmiar wartości 10, zmiennej af wartości 0.22, oraz zostanie wypisana pomoc do pakietu z uwzględnieniem stałej tekstowej help (patrz wyżej).
hydra:/usr/home/bin/> program -N 23 -bez_pc
spowoduje przypisanie zmiennej rozmiar wartości 23, ustawienia pc_flag na TRUE, a zmienna af pozostanie bez zmian, tzn. równa 0.1.
Załóżmy, że mamy już wygenerowany obiekt PC (nieważne, czy wydobyty ze SLESu funkcją SLESGetPC(), czy wygenerowany funkcją PCCreate()) i ustawione operatory. Aby ustawić preconditioner Schwarza, należy wywołać funkcję: PCSetType(pc,PCASM). Funkcje służące do sterowania tym preconditionerem pozwalają na automatyzację tworzenia jego struktury. W zasadzie jedyną rzeczą, jaką użytkownik musi sam zaimplementować, jest dekompozycja obszaru.
Funkcje ASM można podzielić na trzy zasadnicze typy:
Przykład
...
#define MP MPI_COMM_WORLD
IS is;
Int indeksy[4]={0,5,12,17,22};
...
ISCreateGeneral(MP,5, indeksy,&is);
...
W tym przypadku wygenerowany zostanie zbiór indeksów
{0,5,12,17,22}
|
Przykład
... #define MP MPI_COMM_WORLD IS is; ... ISCreateStride(MP,5,2,3,&is); ... W tym przypadku wygenerowany zostanie zbiór indeksów
{2,5,8,11,14} Tak jakby wstawić tablicę wygenerowaną następująco: for(i=0;i<n;i++)indeksy[i]=2+i*3;
|

Dekompozycję obszaru podaje się jako tablicę zbiorów indeksów (IS), gdzie każdy zbiór indeksów podaje numery współrzędnych w jednym podobszarze. Przypuśćmy, że mamy obszar L-kształtny. Rysunek 7, pokazuje jak ponumerowane są węzły siatki. Cała funkcja siatkowa na tym obszarze zapisywana jest jako wektor o 48 współrzędnych. Wprowadzamy dekompozycję na trzy podobszary jak na rysunku 5. Dekompozycję w tym przypadku zapisujemy więc jako 3elementową tablicę zmiennych typu IS. Będą to następujące zbiory:
 {19,..,23,27,...,31,35,...,39,43,...,47
}
{19,..,23,27,...,31,35,...,39,43,...,47
} {16,..,20,24,..28,32..,36,40,...,44}
{16,..,20,24,..28,32..,36,40,...,44} {0,...,19}
{0,...,19}
Przy podawaniu dekompozycji musimy sami zdecydować, czy zdajemy się na funkcję automatycznie ustawiającą wielkość zakładki, czy zakładkę definiujemy sami i takie zbiory indeksów musimy wygenerować. W powyższym przykładzie dekompozycja została zdefiniowana z zakładkami, bo tak uzyskujemy pełną kontrolę nad geometrią podobszarów. Zbiór indeksów wstawiamy do PC przy pomocy funkcji PCASMSetTotalSubdomains(PC pc, int N, IS *is). Pierwszy argument to zmienna typu PC służąca do kontroli obiektu, drugi to całkowita liczba podobszarów a trzeci to tablica zbiorów indeksów opisująca dekompozycję. Ta funkcja automatycznie rozdysponowuje podobszary pomiędzy procesory. Można też samemu zdecydować ile podobszarów będzie w poszczególnych procesorach przy pomocy funkcji PCASMSetLocalSubdomains(PC pc, int n, IS *is) Pierwszy argument to zmienna typu PC powiązana z obiektem, drugi to liczba podobszarów w danym procesorze, a trzeci to tablica zbiorów indeksów z podobszarami dla tego procesora. Ostatnia zmienna może przyjąć wartość PETSC_NULL, gdy PETSc ma samo zdecydować, które podobszary mają trafić do danego procesora. Gdy powyższe funkcje są wywoływane w programie wykonywanym wielowątkowo, to muszą być wywołane przez wszystkie procesy w obrębie komunikatora MPI, przy czym pierwsza musi być wywołana z tą samą we wszystkich wątkach tablicą zbiorów indeksów.
Dodatkowo autorzy dołączyli do pakietu funkcję PCASMCreateSubdomains2D(int m,int n,int M,int N,int dof,int overlap,int *Nsub,IS **is) która służy do utworzenia dekompozycji prostokątnego obszaru dwuwymiarowego z regularną siatką na M*N prostokątnych podobszarów. Argumenty tej funkcji to po kolei: rozmiar siatki w poziomie, rozmiar siatki w pionie, liczba podobszarów w poziomie, liczba podobszarów w pionie, liczba stopni swobody siatki, szerokość zakładki, wskaźnik do zmiennej, w której znajdzie się całkowita liczba utworzonych podobszarów i ostatni argument to wskaźnik do tablicy zbiorów indeksów, która zostanie wygenerowana przez tę funkcję.

Zakładka generowana automatycznie tworzona jest na podstawie niezerowej struktury macierzy. Konkretne problemy mogą prowadzić do innych struktur zakładki, na przykład funkcja PCASMCreateSubdomains2D() tworzy zakładkę w innym kształcie niż zakładka utworzona automatycznie (Rysunek 9), dlatego dobrze jest samemu zdefiniować dekompozycję wraz z zakładkami.
PC_ASM_BASIC metoda podstawowa pełna interpolacja i restrykcja
PC_ASM_RESTRICT pełna restrykcja, lokalna interpolacja w procesorze
PC_ASM_INTERPOLATE pełna interpolacja, lokalna restrykcja w procesorze
PC_ASM_NONE lokalna restrykcja i interpolacja w procesorze
Wszystkie te metody związane są ściśle z implementacją równoległą, to znaczy wszelkie obcięcia i interpolacje odnoszą się do danych pochodzących z innych procesorów. Jeżeli jeden procesor ma kilka podobszarów to w obrębie jego danych stosowana jest zawsze metoda podstawowa - i słusznie, bo wszystkie innowacje mają na celu wyeliminowanie zbędnej komunikacji, która często jest kosztowna, stąd w obrębie jednego procesora nie warto stosować tych metod. Wszystkie metody rozwiązują układ bez grubej siatki.
#include "sles.h";
PC pc,pc1;
SLES *slesy;
...
PCASMGetSubSLES(pc,&n,&zero,&slesy);
for(i=zero;i<zero+n;i++)
{
SLESGetPC(slesy[i],&pc1);
PCSetType(pc1,PCICC);
}
 Rysunek
10. Struktura preconditionera Schwarza dla 4 podobszarów.
Rysunek
10. Struktura preconditionera Schwarza dla 4 podobszarów.Podobnie będzie się nam pracowało z kolejnym składnikiem PETSc, służącym tym razem do (równoległego) rozwiązywania nieliniowych układów równań: SNES. Zanim przejdziemy do omówienia sposobów wykorzystania tego komponentu PETSc, zrobimy najpierw krótki wstęp o metodach rozwiązywania równań nieliniowych. Po przypomnieniu metod rozwiązywania takich równań, przedstawimy ogólny schemat użycia SNESu - nieliniowego analogonu SLESu - obiektu służącego do rozwiązywania równań nieliniowych. Następnie zobaczymy na dwóch przykładach, jak w praktyce wygląda stosowanie SNESu. Między innymi, użyjemy SNESu do zaimplementowania prostego programu rozwiązującego układ powstały po dyskretyzacji nieliniowego zagadnienia konwekcji-dyfuzji

W trakcie szczegółowego omawiania przedstawionych przykładów, poznamy techniki efektywnego wykorzystania całej naszej wiedzy o dotychczas poznanych składnikach PETSc (macierzach, SLESie, itp.) oraz omówimy bliżej bardzo pożyteczną ideę kontekstu.
Zakończymy tę część opisem możliwości modyfikacji działania SNESu: wyboru wariantu metody Newtona, kryteriów stopu, itd. oraz podsumowaniem składni wywołań procedur SNESu.

przy czym rozważymy przypadek gdy  .
.
Jak wiemy, dyskretyzacja taka prowadzi do następującej postaci macierzowej (patrz [6]):
gdzie macierz A ma postać:
 ,
, 
static char help[] = "Rozwiązuje pięciodiagonalny układ z użyciem SLES.\n\n";
#include "sles.h"
#include <stdio.h>
int main(int argc,char **args)
{
Vec x, b, u;
Mat A; /* macierz układu równań */
SLES sles; /* kontekst SLESu */
int ierr, i, n = 10, m = 10,col[5], its,flg,k,l;
Scalar minus_one = -1.0, one = 1.0, val[5],h;
double norm;
PetscInitialize(&argc,&args,PETSC_NULL,help);
OptionsGetInt(PETSC_NULL,"n",&n,&flg);
OptionsGetInt(PETSC_NULL,"m",&m,&flg);
/* Tworzenie wektorów */
VecCreate(MPI_COMM_WORLD,PETSC_DECIDE,n*m,&x);
VecDuplicate(x,&b);
VecDuplicate(x,&u);
/* Wartości wektora prawej strony */
val[0]=1;
VecSet(val,b);
/* Tworzenie macierzy laplasjanu na prostokacie mxn */
MatCreate(MPI_COMM_WORLD,n*m,n*m,&A);
h=1/(m*n+1);
val[0] = -1.0/h; val[1] = -1.0/h; val[2] = 4.0/h;
val[3] = -1.0/h; val[4] = -1.0/h;
for (i=0; i<n*m; i++ )
{
col[0]=i+1; col[1]=i-1; col[2]=i;
col[3]=i+m; col[4]=i-m;
l=0;k=5;
if(i<m) k=4;
if((i+1)>(n-1)*m){k=4;col[3]=i-m;}
if((i%m)==0){l=1;k=k-1;col[1]=i+1;}
if((i+1)%m==0)if(i!=0){l=1;k=k-1;}
MatSetValues(A,1,&i,k,&col[l],&val[l],INSERT_VALUES);
}
MatSetValues(A,1,&i,k,&col[l],&val[l],INSERT_VALUES);
MatAssemblyBegin(A,MAT_FINAL_ASSEMBLY);
MatAssemblyEnd(A,MAT_FINAL_ASSEMBLY);
/* Tworzenie kontekstu SLES */
SLESCreate(MPI_COMM_WORLD,&sles);
/*Ustawienie operatorów */
SLESSetOperators(sles,A,A,DIFFERENT_NONZERO_PATTERN);
SLESSetFromOptions(sles);
/* rozwiązanie układu liniowego */
SLESSolve(sles,b,x,&its);
/* Sprawdzenie błedu */
VecAXPY(&minus_one,u,x);
VecNorm(x,NORM_2,&norm);
PetscPrintf(MPI_COMM_WORLD,"Norma błędu %g, %d iteracji\n",norm,its);
/* Zwolnienie przestrzeni roboczej */
VecDestroy(x);VecDestroy(u);
VecDestroy(b);MatDestroy(A);
SLESDestroy(sles);
PetscFinalize();
return(0);
}
hydra:/usr/home/students/bojko/> przyklad Norma błędu 0.00893566, 65 iteracji hydra:/usr/home/students/bojko/>

|
Jeżeli chcemy wybrać metodę z poziomu kodu źródłowego, trzeba to zrobić w następujący sposób: zadeklarować zmienną typu KSP i powiązać ją z obiektem KSP ze SLESu funkcją SLESGetKSP(SLES sles,KSP *ksp), a potem użyć funkcji KSPSetType(KSP ksp, KSPType itmethod). Zmienna sles jest związana z obiektem SLES, z którego wydobywamy obiekt KSP, z kolei *ksp to wskaźniki do obiektu KSP. itmethod jest stałą PETSc oznaczającą rodzaj metody. Spis stałych znajduje się w tabelce poniżej. Jeżeli po ustawieniu rodzaju metody z poziomu kodu źródłowego funkcją KSPSetType() wywołamy funkcję SLESSetFromOptions() to dodatkowo będziemy w stanie zmienić metodę opcją z linii komend, ale domyślną metodą będzie metoda ustawiona z poziomu kodu źródłowego.
Można też wybrać metodę uruchamiając program z opcją -ksp_type <metoda>. Poniższa tabelka podaje wszystkie stałe, predefiniowane w PETSc i opcje, którymi wybieramy metodę z linii komend
|
|
|
|
| KSPRICHARDSON | richardson | Normalny |
| KSPCHEBYSHEV | chebyshev | Normalny |
| KSPCG | cg | Normalny |
| KSPGMRES | gmres | Przewarunkowany |
| KSPTCQMR | tcqmr | Przewarunkowany |
| KSPBCGS | bcgs | Przewarunkowany |
| KSPCGS | cgs | Przewarunkowany |
| KSPTFQMR | tfqmr | Przewarunkowany |
| KSPCR | cr | Przewarunkowany |
| KSPLSQR | lsqr | Przewarunkowany |
| KSPPREONLY | preonly | Przewarunkowany |
#include "sles.h"
int main(int argc,char **args)
{
Vec x,b;
SLES sles;
KSP ksp;
int its;
/* inicjalizacja */
PetscInitialize(&argc,&args,PETSC_NULL,PETSC_NULL);
SLESCreate(MPI_COMM_WORLD,&sles);
/* wybór metody KSP */
SLESGetKSP(sles,&ksp);
KSPSetType(ksp,KSPRICHARDSON);
/* W tej części programu muszą zostać ustawone wszystkie parametry SLESu, */
/* omówione w poprzednich rozdziałach. Dla większej przejrzystości */
/* przykładu zostały pominięte */
SLESSetFromOptions(sles);
SLESSolve(sles,x,b,&its);
/* zakończenie */
SLESDestroy(sles);
PetscFinalize();
}


 (w tym przypadku stwierdzona
zostaje rozbieżność)
(w tym przypadku stwierdzona
zostaje rozbieżność)KSPSetTolerances(KSP ksp,double rtol,double atol,double dtol,int maxits).
rtol = 10-5, atol = 10-50, dtol = 105 i maxits = 105.
Inne przydatne opcje dla KSP to -ksp_monitor i -ksp_xmonitor, które na bieżąco śledzą (odpowiednio: w trybie tekstowym lub graficznym) przebieg procesu iteracyjnego rozwiązywania równania. W pierwszym przypadku na każdym kroku zostaje wypisana na konsolę norma wektora residualnego. W drugim przypadku przebieg zbieżności obserwujemy w oknie graficznym.
 |
 |
|
|
|
Po wywołaniu funkcji SLESSolve() należy wywołać funkcję KSPComputeExtremeSingularValues(KSP ksp, double *emax, double *emin), która podaje najmniejszą i największą wartość własną operatora użytego w konkretnej metodzie iteracyjnej.
KSP ksp; double emax,emin; ... SLESGetKSP(sles,&ksp); KSPSetMethod(ksp,KSPCG); KSPSetComputeSingularValues(ksp); SLESSolve(sles); KSPComputeExtremeSingularValues(ksp,&emax,&emin); PetscPrintf(MPI_COMM_WORLD,"Uwarunkowanie operatora wynosi %d\n",emax/emin); ...
 Rysunek
6 Wynik działania opcji -ksp_plot_eigenvalues.
Rysunek
6 Wynik działania opcji -ksp_plot_eigenvalues.Można także wykorzystać opcję PETSc -ksp_plot_eigenvalues, która powinna wyświetlić wyliczony w/w metodami przybliżony rozkład wartości własnych macierzy naszego układu.

polega na zastąpieniu tego układu równoważnym, mnożąc układ wyjściowy przez odwrotność pewnej macierzy B, zwanej preconditionerem:
 .
.Jeżeli teraz zaczniemy rozwiązywać powyższy układ metodą iteracyjną, np. metodą iteracji prostej to otrzymujemy następujące wzory na kolejne przybliżenia rozwiązania:
 .
.Oznaczając  mamy:
mamy:
 ,
,czyli w każdym kroku iteracyjnym mnożymy wektor residualny przez odwrotność macierzy B: po prostu rozwiązujemy układ z macierzą preconditionera. Od macierzy B żądamy:
 :
jak wiadomo, prędkość zbieżności metod iteracyjnych zazwyczaj silnie zależy
do współczynnika uwarunkowania macierzy układu; zmniejszenie tego współczynnika
poważnie wpływa na obniżenie liczby iteracji i tym samym - kosztu rozwiązania
całego układu równań; szczegóły można znaleźć w [6].
:
jak wiadomo, prędkość zbieżności metod iteracyjnych zazwyczaj silnie zależy
do współczynnika uwarunkowania macierzy układu; zmniejszenie tego współczynnika
poważnie wpływa na obniżenie liczby iteracji i tym samym - kosztu rozwiązania
całego układu równań; szczegóły można znaleźć w [6].Do kontroli preconditionera służy obiekt typu PC, który jest częścią obiektu KSP. Intuicyjnie wydaje się to oczywiste: konkretna metoda iteracyjna praktycznie zawsze musi być stosowana z preconditionerem, na przykład możemy wybrać metodę sprzężonych gradientów z preconditionerem Schwarza. W tym rozdziale zostaną omówione podstawowe zasady korzystania z PC, następnie typy preconditionerów zawarte w pakiecie oraz, dokładnie, metoda tworzenia własnych preconditionerów.
W świetle tego co przed chwilą stwierdziliśmy, każdy utworzony obiekt typu SLES zawiera już obiekt PC. Aby uzyskać do niego bezpośredni dostęp należy skojarzyć go ze zmienną typu PC przy pomocy funkcji SLESGetPC(SLES sles,PC *pc);
SLES sles; PC pc; ... SLESCreate(MPI_COMM_WORLD,&sles); SLESGetPC(sles,&pc); ...
Najczęściej wykorzystywaną operacją na PC jest PCSetType(PC pc,PCType type) - funkcja ustawiająca typ preconditionera. Pierwszy argument to obiekt PC dla którego ustawiamy typ, drugim jest jeden z predefiniowanych typów:
|
|
|
|
| Jacobi | PCJACOBI | jacobi |
| Blokowy Jacobi | PCBJACOBI | bjacobi |
| Blokowy GaussSeidel (tylko sekwencyjny) | PCBGS | bgs |
| SOR (i SSOR) | PCSOR | sor |
| SOR z trickiem Eisenstat'a | PCEISENSTAT | eisenstat |
| Niekompletny rozkład Choleskiego | PCICC | icc |
| Niekompletny rozkład LU | PCILU | ilu |
| Addytywna metoda Schwarza | PCASM | asm |
| eliminacja Gaussa LU | PCLU | lu |
| Bez preconditionera | PCMINUS_ONE | minus_one |
| Zdefiniowany przez użytkownika | PCSHELL | shell |
SLES sles; KSP ksp; PC pc; ... SLESCreate(MPI_COM_WORLD,&sles); ... SLESGetKSP(sles,&ksp); KSPSetType(ksp,KSPPREONLY); SLESGetPC(sles,&pc); PCSetType(pc,LU); ... SLESSolve(sles);
... SLESCreate(MPI_COMM_WORLD,&sles); SLESGetPC(sles,&pc); PCSetType(pc,PCICC); ...
|
|
|
| PCASMGetSubSLES() | Wydobywa z preconditionera tablicę SLESów służących do rozwiązywania zadania w podobszarach. |
| PCASMSetLocalSubdomains() | Ustawia ile obszarów obsługuje procesor wywołujący tę funkcję. |
| PCASMSetOverlap() | Ustawia szerokość zakładki między obszarami. |
| PCASMSetTotalSubdomains() | Ustawia całkowitą liczbę podobszarów. |
| PCASMSetType() | Ustawia rodzaj metody Schwarza - sposób w jaki dane będą przekazywane między procesorami. |
Tak więc, aby wstawić do SLESu własny preconditioner należy:
przyklad8_1.c
#include "common.h"
static char help[] ="Preconditioner trywialny\n";
/*************************************************
Działanie preconditionera
*************************************************/
int Pierwszy_pc(void *ctx,Vec v0,Vec v1)
{
VecCopy(v0,v1);
return(0);
}
/*************************************************
Program glowny
*************************************************/
int main(int argc,char **args)
{
PetscInitialize(&argc,&args,PETSC_NULL,help);
Przygotuj_zadanie(N,&sles,&A,&u0,&u,&f);
/*ustawienie preconditionera powłokowego */
SLESGetPC(sles,&pc);
PCSetType(pc,PCSHELL);
PCShellSetApply(pc,Pierwszy_pc,PETSC_NULL);
PCShellSetName(pc,"Mój pierwszy preconditioner");
/*rozwiązanie zadania */
Rozwiaz(sles);
/*Zwalnianie pamięci*/
Zwolnij_pamiec(sles,A,u,u0,f);
PetscFinalize();
}
Po uruchomieniu mamy:
hydra:/usr/home/bin/> przyklad Iteracje = 351 Blad = 6.364062e-04 hydra:/usr/home/bin/>
 jest w tym wypadku macierzą identyczności.
jest w tym wypadku macierzą identyczności. ,
,dla odpowiednio dobranej wartości parametru  .
Jak wiadomo, najlepszy parametr metody Richardsona uzyskuje się, korzystając
z najmniejszej i największej wartości własnej macierzy (opis metody Richardsona
można znaleźć w [6]). Dlatego jako
kontekst podane zostaje oszacowanie na spektrum macierzy. Podobnie jak
w poprzednim przypadku, pliki common.h i common.c są
podane w przykładzie podsumowującym na stronie 37.
.
Jak wiadomo, najlepszy parametr metody Richardsona uzyskuje się, korzystając
z najmniejszej i największej wartości własnej macierzy (opis metody Richardsona
można znaleźć w [6]). Dlatego jako
kontekst podane zostaje oszacowanie na spektrum macierzy. Podobnie jak
w poprzednim przypadku, pliki common.h i common.c są
podane w przykładzie podsumowującym na stronie 37.
przyklad8_2.c
#include "common.h"
static char help[] ="Preconditioner Richardsona\n";
/***************************************************
Kontekst dla preconditionera
***************************************************/
typedef struct {
double lambdamin;
double lambdamax;} Kontekst;
Kontekst k_pc;
/***************************************************
Dzialanie preconditionera
***************************************************/
int Richardson_pc(void *ctx,Vec v0,Vec v1)
{
Scalar val=1;
Kontekst *kn=(Kontekst*)ctx;
val=2/(kn->lambdamin+kn->lambdamax);
VecCopy(v0,v1);
VecScale(&val,v1);
return(0);
}
/**************************************************
Program glowny
**************************************************/
main(int argc,char **args)
{
PetscInitialize(&argc,&args,PETSC_NULL,help);
Przygotuj_zadanie(N,&sles,&A,&u0,&u,&f);
SLESGetPC(sles,&pc);
PCSetType(pc,PCSHELL);
PCShellSetApply(pc,Richardson_pc,(void*)&k_pc);
PCShellSetName(pc,"Preconditioner Richardsona");
k_pc.lambdamin=1/N;
k_pc.lambdamax=1;
Rozwiaz(sles);
Zwolnij_pamiec(sles,A,u0,u,f);
PetscFinalize();
}
Po uruchomieniu mamy:
hydra:/usr/home/bin/> przyklad Iteracje = 80 Blad = 4.366761e-04 hydra:/usr/home/bin/>

gdzie aii oznaczają elementy diagonalne macierzy zadania A. W implementacji tego preconditionera skorzystamy więc ze zmiennej kontekstowej: jako kontekst podamy główną diagonalę macierzy zadania. Zwróćmy uwagę na funkcję PrzygotujPreconditioner() - wykonuje on podobną pracę, co w standardowym przypadku funkcja PCSetUp().
przyklad8_3.c
#include "common.h"
static char help[] ="Preconditioner Jacobi'ego";
/***********************************************
Inicjalizacja kontekstu preconditionera
***********************************************/
int PrzygotujPreconditioner(Mat A,Vec *k_pc)
{
int m,n;
MatGetSize(A,&m,&n);
If(m!=n)
{
PetscPrintf(MPI_COMM_WORLD,"Hej!! Macierz nie jest kwadratowa\n");
}
VecCreate(MPI_COMM_WORLD,PETSC_DECIDE,m,k_pc);
MatGetDiagonal(A,(*k_pc));
return(0);
}
/***********************************************
Dzialanie preconditionera
***********************************************/
int wlasny_pc(void *ctx,Vec v0,Vec v1)
{
Vec *k_pc=(Vec*)ctx;
/* Dzialanie preconditionerem Jacobiego */
VecPointwiseDivide(v0,*k_pc,v1);
return(0);
}
/***********************************************
Program glowny
***********************************************/
int main(int argc,char **args)
{
Vec diag;
PetscInitialize(&argc,&args,PETSC_NULL,help);
Przygotuj_zadanie(N,&sles,&A,&u0,&u,&f);
SLESGetPC(sles,&pc);
PCSetType(pc,PCSHELL);
PrzygotujPreconditioner(A,&diag);
PCShellSetApply(pc,wlasny_pc,&diag);
PCShellSetName(pc,"Preconditioner Jacobi'ego");
Rozwiaz(sles);
PetscFinalize();
}
Po uruchomieniu mamy:
hydra:/usr/home/bin/> przyklad Iteracje = 63 Blad = 4.364761e-04 hydra:/usr/home/bin/>
przyklad8_4.c
#include "common.h"
static char help[] ="Addytywny preconditioner Schwarza z grubą siatką
Marcin Bojko";
/* Tutaj definiujemy strukture, która bedzie kontekstem preconditionera */
typedef struct {
Mat I,Ag; /* macierz interpolacji i zadania na grubej siatce */
int rozmiar,mg; /* mg to rozmiar grubej siatki*/
int m,M,zakl; /* M to ilość podobszarów w jednym kierunku */
/* wszystkich podobszarów będzie M*M */
IS *boxy; /* tu będzie składowana dekompozycja */
Vec fg,ug; /* pomocnicze wektory zadania na grubej siatce */
Scalar value[3],val;
SLES slesg; /* SLES dla zadania na gubej siatce */
PC pc_box; /* preconditioner Schwarza na boxach */
} asm_ctx;
int Preconditioner(void *pointer,Vec u0,Vec u1);
void Przygotuj_macierz_interpolacji(asm_ctx *ctx)
{
int i,j,row,col,r,k,l;
int mg=ctx->mg,m=ctx->m,rozmiar=ctx->rozmiar;
Scalar val;
MatCreate(MPI_COMM_WORLD,rozmiar*rozmiar,mg*mg,&ctx->I);
for(j=0;j<mg;j++)
for(i=0;i<mg;i++)
{
col=i+mg*j;
row=r=(m-1)*rozmiar+m-1+i*m+j*m*rozmiar;
val=1;
MatSetValues(ctx->I,1,&row,1,&col,&val,INSERT_VALUES);
for(k=1;k<m;k++)
{
val=(Scalar)(m-k)/m;
for(l=0;l<6*k;l++)
{
if((l>1)&&(l<k))row=r-k*rozmiar+l;
if((l>k-1)&&(l<2*k))row=r-k*rozmiar+k+(l-k)*rozmiar;
if((l>2*k-1)&&(l<3*k))row=r+k+(l-2*k)*(rozmiar-11);
if((l>3*k-1)&&(l<4*k))row=r+k*rozmiar-(l-3*k);
if((l>4*k-1)&&(l<5*k))row=r+k*(rozmiar-1)-(l-4*k)*rozmiar;
if((l>5*k-1)&&(l<6*k))row=r-k-(l-5*k)*(rozmiar-1);
MatSetValues(ctx->I,1,&row,1,&col,&val,INSERT_VALUES);
}
}
}
MatAssemblyBegin(ctx->I,MAT_FINAL_ASSEMBLY);
MatAssemblyEnd(ctx->I,MAT_FINAL_ASSEMBLY);
MatCompress(ctx->I);
}
Przygotuj_preconditioner(asm_ctx *ctx,PC *pc_shell)
{
int flag;
int rozmiar=ctx->rozmiar,M=ctx->M;
int zakl=ctx->zakl,mg=ctx->mg;
PC pcg;
KSP ksp;
/*Przygotowanie preconditionera Schwarza */
PCCreate(MPI_COMM_WORLD,&ctx->pc_box);
PCSetType(ctx->pc_box,PCASM);
PCASMCreateSubdomains2D(rozmiar,rozmiar,M,M,1,zakl,&i,&ctx->boxy);
PCASMSetLocalSubdomains(ctx->pc_box,i,ctx->boxy);
PCSetOperators(ctx->pc_box,A,A,SAME_PRECONDITIONER);
PCSetVector(ctx->pc_box,u);
PCASMSetOverlap(ctx->pc_box,0);
PCSetUp(ctx->pc_box);
/* gruba siatka */
VecCreate(MPI_COMM_WORLD,PETSC_DECIDE,mg*mg,&ctx->fg);
VecDuplicate(ctx->fg,&ctx->ug);
Przygotuj_macierz_laplasjanu(&ctx->Ag,mg,mg);
SLESCreate(MPI_COMM_WORLD,&ctx->slesg);
SLESSetOperators(ctx->slesg,ctx->Ag,ctx->Ag,SAME_PRECONDITIONER);
SLESGetPC(ctx->slesg,&pcg);
PCSetType(pcg,PCLU);
SLESGetKSP(ctx->slesg,&ksp);
KSPSetType(ksp,KSPPREONLY);
Przygotuj_macierz_interpolacji(ctx);
}
int main(int argc,char **args)
{
KSP ksp;
PC pc_shell;
asm_ctx ams;
double norma,emin,emax;
int i,flag;
PetscInitialize(&argc,&args,PETSC_NULL,help);
ams.M=4;
ams.mg=7;
ams.zakl=1;
ams.m=8;
OptionsGetInt(PETSC_NULL,"M",&ams.M,&flag);
OptionsGetInt(PETSC_NULL,"mg",&ams.mg,&flag);
OptionsGetInt(PETSC_NULL,"m",&ams.m,&flag);
OptionsGetInt(PETSC_NULL,"zakl",&ams.zakl,&flag);
ams.rozmiar=(ams.mg+1)*ams.m-1;
Przygotuj_zadanie(ams.rozmiar,&sles,&A,&u0,&u,&f);
SLESGetKSP(sles,&ksp);
SLESGetPC(sles,&pc_shell);
Przygotuj_preconditioner(&ams,&pc_shell);
PCSetType(pc_shell,PCSHELL);
PCShellSetApply(pc_shell,Preconditioner,&ams);
PCShellSetName(pc_shell,"asmbox");
KSPSetType(ksp,KSPGMRES);
KSPSetComputeSingularValues(ksp);
SLESSetFromOptions(sles);
Rozwiaz(sles);
KSPComputeExtremeSingularValues(ksp,&emax,&emin);
PetscFPrintf(MPI_COMM_WORLD,stderr," Uwarunkowanie = %e\n\n",emax/emin);
SLESDestroy(ams.slesg);
MatDestroy(ams.Ag);
VecDestroy(ams.ug);
VecDestroy(ams.fg);
MatDestroy(ams.I);
Zwolnij_pamiec(sles,A,u0,u,f);
PetscFinalize();
}
int Preconditioner(void *pointer,Vec v0 ,Vec v1)
{
asm_ctx *ams=(asm_ctx*)pointer;
int its;
/*rozwiązanie na boxach */
PCApply(ams->pc_box,v0,v1);
/* rozwiazanie na grubej siatce */
MatMultTrans(ams->I,v0,ams->fg);
SLESSolve(ams->slesg,ams->fg,ams->ug,&its);
VecScale(&val,ams->ug);
MatMultAdd(ams->I,ams->ug,v1,v1);
return(0);
}
hydra:/usr/home/bin/> przyklad8_4 Liczba iteracji = 24 Blad = 6.442081e-04 Uwarunkowanie = 5.804242e+01 hydra:/usr/home/bin/>
Poniżej przytaczamy pliki źródłowe common.c i common.h, z których korzystaliśmy w niniejszym rozdziale.
common.h #include "sles.h" Vec u,u0,f; Mat A; SLES sles; PC pc; int N=100,its,i; Scalar val; double norma; int Rozwiaz(SLES sles); int Przygotuj_zadanie(int rozmiar,SLES *sles,Mat *A,Vec *u0,Vec *u,Vec *f); int Zwolnij_pamiec(SLES sles,Mat A,Vec u0,Vec u,Vec f); int Przygotuj_macierz_laplasjanu(Mat *A,int roz_x,int roz_y);
common.c #include "common.h" int Przygotuj_macierz_laplasjanu(Mat *A,int roz_x,int roz_y) { Scalar val; int n,m,i,j; n=roz_x*roz_y; MatCreateSeqAIJ(MPI_COMM_WORLD,n,n,5,PETSC_NULL,A);; for(i=0;i<n;i++) { val=4; MatSetValues(*A,1,&i,1,&i,&val,INSERT_VALUES);; /* elementy na dalekich kodiagonalach */ val=-1; j=i+roz_y; if(j<n) { MatSetValues(*A,1,&i,1,&j,&val,INSERT_VALUES);; MatSetValues(*A,1,&j,1,&i,&val,INSERT_VALUES);; } /* elementy na bliskich kodiagonalach */ j=i+1; val=-1; if(j<n)if(((i+1)%roz_x)!=0||(i==0)) { MatSetValues(*A,1,&i,1,&j,&val,INSERT_VALUES);; MatSetValues(*A,1,&j,1,&i,&val,INSERT_VALUES);; } } MatAssemblyBegin(*A,MAT_FINAL_ASSEMBLY); ; MatAssemblyEnd(*A,MAT_FINAL_ASSEMBLY); ; MatCompress(*A); return(0); } /*Przygotowanie zadania testowego */ int Przygotuj_zadanie_testowe(int N,SLES *sles,Mat *A,Vec *u0,Vec *u,Vec *f) { PetscRandom rctx; VecCreate(MPI_COMM_WORLD,PETSC_DECIDE,N*N,f); VecDuplicate(*f,u); VecDuplicate(*f,u0); Przygotuj_macierz_laplasjanu(A,N,N); SLESCreate(MPI_COMM_WORLD,sles); SLESSetOperators(*sles,*A,*A,SAME_PRECONDITIONER); PetscRandomCreate(MPI_COMM_WORLD,RANDOM_DEFAULT,&rctx); VecSetRandom(rctx,*u0); PetscRandomDestroy(rctx); MatMult(*A,*u0,*f); return(0); } /* Zwolnienie pamięci po zadaniu testowym */ int Zniszcz_zadanie_testowe(SLES sles,Mat A,Vec u0,Vec u,Vec f) { SLESDestroy(sles); VecDestroy(u0); VecDestroy(u); VecDestroy(f); MatDestroy(A); } int Rozwiarz(SLES sles) { SLESSetFromOptions(sles); SLESSolve(sles,f,u,&its); /*Podanie wynikow*/ val=-1; VecAXPY(&val,u,u0); VecNorm(u0,NORM_2,&norma); PetscFPrintf(MPI_COMM_WORLD,stderr,"\nLiczba iteracji = %i\n",its); PetscFPrintf(MPI_COMM_WORLD,stderr," Blad = %e\n",(double)norma); }
BOPT=O include $(PETSC_DIR)/bmake/$(PETSC_ARCH)/base CFLAG = $(CPPFLAGS) D__SDIR__='"$(LOCDIR)"' $(BS_INCLUDE) LIBBASE = libpetscsles LOCDIR = /home/students/bojko/source/ wszystko: przykład8_1 przykład8_2 przykład8_3 przykład8_4 przykład8_1: przyklad8_1.o $(CLINKER) -o przyklad8_1 przyklad8_1.o common.o $(PETSC_LIB) $(RM) przyklad8_1.o przyklad8_2: przyklad8_2.o $(CLINKER) -o przyklad8_2 przyklad8_2.o common.o $(PETSC_LIB) $(RM) przyklad8_2.o przyklad8_3: przyklad8_3.o $(CLINKER) -o przyklad8_3 przyklad8_3.o common.o $(PETSC_LIB) $(RM) przyklad8_3.o przyklad8_4: przyklad8_4.o $(CLINKER) -o przyklad8_4 przyklad8_4.o common.o $(PETSC_LIB) $(RM) przyklad8_4.o
hydra:/usr/home/students/bojko/>make przyklad8_1
Plik wykonywalny otrzyma nazwę przyklad8_1, więc będziemy go uruchamiać, pisząc
hydra:/usr/home/students/bojko/>przyklad8_1
Można też od razu skompilować wszystkie programy (pod warunkiem, że wszystkie pliku źródłowe są w jednym katalogu) poleceniem:
hydra:/usr/home/students/bojko/>make wszystko
Załóżmy, że mamy już wygenerowany obiekt PC (nieważne, czy wydobyty ze SLESu funkcją SLESGetPC(), czy wygenerowany funkcją PCCreate()) i ustawione operatory. Aby ustawić preconditioner Schwarza, należy wywołać funkcję: PCSetType(pc,PCASM). Funkcje służące do sterowania tym preconditionerem pozwalają na automatyzację tworzenia jego struktury. W zasadzie jedyną rzeczą, jaką użytkownik musi sam zaimplementować, jest dekompozycja obszaru.
Funkcje ASM można podzielić na trzy zasadnicze typy:
Przykład
...
#define MP MPI_COMM_WORLD
IS is;
Int indeksy[4]={0,5,12,17,22};
...
ISCreateGeneral(MP,5, indeksy,&is);
...
W tym przypadku wygenerowany zostanie zbiór indeksów
{0,5,12,17,22}
|
Przykład
... #define MP MPI_COMM_WORLD IS is; ... ISCreateStride(MP,5,2,3,&is); ... W tym przypadku wygenerowany zostanie zbiór indeksów
{2,5,8,11,14} Tak jakby wstawić tablicę wygenerowaną następująco: for(i=0;i<n;i++)indeksy[i]=2+i*3;
|

Dekompozycję obszaru podaje się jako tablicę zbiorów indeksów (IS), gdzie każdy zbiór indeksów podaje numery współrzędnych w jednym podobszarze. Przypuśćmy, że mamy obszar L-kształtny. Rysunek 7, pokazuje jak ponumerowane są węzły siatki. Cała funkcja siatkowa na tym obszarze zapisywana jest jako wektor o 48 współrzędnych. Wprowadzamy dekompozycję na trzy podobszary jak na rysunku 5. Dekompozycję w tym przypadku zapisujemy więc jako 3elementową tablicę zmiennych typu IS. Będą to następujące zbiory:
 {19,..,23,27,...,31,35,...,39,43,...,47
}
{19,..,23,27,...,31,35,...,39,43,...,47
} {16,..,20,24,..28,32..,36,40,...,44}
{16,..,20,24,..28,32..,36,40,...,44} {0,...,19}
{0,...,19}
Przy podawaniu dekompozycji musimy sami zdecydować, czy zdajemy się na funkcję automatycznie ustawiającą wielkość zakładki, czy zakładkę definiujemy sami i takie zbiory indeksów musimy wygenerować. W powyższym przykładzie dekompozycja została zdefiniowana z zakładkami, bo tak uzyskujemy pełną kontrolę nad geometrią podobszarów. Zbiór indeksów wstawiamy do PC przy pomocy funkcji PCASMSetTotalSubdomains(PC pc, int N, IS *is). Pierwszy argument to zmienna typu PC służąca do kontroli obiektu, drugi to całkowita liczba podobszarów a trzeci to tablica zbiorów indeksów opisująca dekompozycję. Ta funkcja automatycznie rozdysponowuje podobszary pomiędzy procesory. Można też samemu zdecydować ile podobszarów będzie w poszczególnych procesorach przy pomocy funkcji PCASMSetLocalSubdomains(PC pc, int n, IS *is) Pierwszy argument to zmienna typu PC powiązana z obiektem, drugi to liczba podobszarów w danym procesorze, a trzeci to tablica zbiorów indeksów z podobszarami dla tego procesora. Ostatnia zmienna może przyjąć wartość PETSC_NULL, gdy PETSc ma samo zdecydować, które podobszary mają trafić do danego procesora. Gdy powyższe funkcje są wywoływane w programie wykonywanym wielowątkowo, to muszą być wywołane przez wszystkie procesy w obrębie komunikatora MPI, przy czym pierwsza musi być wywołana z tą samą we wszystkich wątkach tablicą zbiorów indeksów.
Dodatkowo autorzy dołączyli do pakietu funkcję PCASMCreateSubdomains2D(int m,int n,int M,int N,int dof,int overlap,int *Nsub,IS **is) która służy do utworzenia dekompozycji prostokątnego obszaru dwuwymiarowego z regularną siatką na M*N prostokątnych podobszarów. Argumenty tej funkcji to po kolei: rozmiar siatki w poziomie, rozmiar siatki w pionie, liczba podobszarów w poziomie, liczba podobszarów w pionie, liczba stopni swobody siatki, szerokość zakładki, wskaźnik do zmiennej, w której znajdzie się całkowita liczba utworzonych podobszarów i ostatni argument to wskaźnik do tablicy zbiorów indeksów, która zostanie wygenerowana przez tę funkcję.

Zakładka generowana automatycznie tworzona jest na podstawie niezerowej struktury macierzy. Konkretne problemy mogą prowadzić do innych struktur zakładki, na przykład funkcja PCASMCreateSubdomains2D() tworzy zakładkę w innym kształcie niż zakładka utworzona automatycznie (Rysunek 9), dlatego dobrze jest samemu zdefiniować dekompozycję wraz z zakładkami.
PC_ASM_BASIC metoda podstawowa pełna interpolacja i restrykcja
PC_ASM_RESTRICT pełna restrykcja, lokalna interpolacja w procesorze
PC_ASM_INTERPOLATE pełna interpolacja, lokalna restrykcja w procesorze
PC_ASM_NONE lokalna restrykcja i interpolacja w procesorze
Wszystkie te metody związane są ściśle z implementacją równoległą, to znaczy wszelkie obcięcia i interpolacje odnoszą się do danych pochodzących z innych procesorów. Jeżeli jeden procesor ma kilka podobszarów to w obrębie jego danych stosowana jest zawsze metoda podstawowa - i słusznie, bo wszystkie innowacje mają na celu wyeliminowanie zbędnej komunikacji, która często jest kosztowna, stąd w obrębie jednego procesora nie warto stosować tych metod. Wszystkie metody rozwiązują układ bez grubej siatki.
#include "sles.h";
PC pc,pc1;
SLES *slesy;
...
PCASMGetSubSLES(pc,&n,&zero,&slesy);
for(i=zero;i<zero+n;i++)
{
SLESGetPC(slesy[i],&pc1);
PCSetType(pc1,PCICC);
}
 Rysunek
10. Struktura preconditionera Schwarza dla 4 podobszarów.
Rysunek
10. Struktura preconditionera Schwarza dla 4 podobszarów.Podobnie będzie się nam pracowało z kolejnym składnikiem PETSc, służącym tym razem do (równoległego) rozwiązywania nieliniowych układów równań: SNES. Zanim przejdziemy do omówienia sposobów wykorzystania tego komponentu PETSc, zrobimy najpierw krótki wstęp o metodach rozwiązywania równań nieliniowych. Po przypomnieniu metod rozwiązywania takich równań, przedstawimy ogólny schemat użycia SNESu - nieliniowego analogonu SLESu - obiektu służącego do rozwiązywania równań nieliniowych. Następnie zobaczymy na dwóch przykładach, jak w praktyce wygląda stosowanie SNESu. Między innymi, użyjemy SNESu do zaimplementowania prostego programu rozwiązującego układ powstały po dyskretyzacji nieliniowego zagadnienia konwekcji-dyfuzji

W trakcie szczegółowego omawiania przedstawionych przykładów, poznamy techniki efektywnego wykorzystania całej naszej wiedzy o dotychczas poznanych składnikach PETSc (macierzach, SLESie, itp.) oraz omówimy bliżej bardzo pożyteczną ideę kontekstu.
Zakończymy tę część opisem możliwości modyfikacji działania SNESu: wyboru wariantu metody Newtona, kryteriów stopu, itd. oraz podsumowaniem składni wywołań procedur SNESu.
 ,
,gdzie  .
Do rozwiązywania równań powyższej postaci używa się metod iteracyjnych,
takich jak metoda iteracji prostej Banacha:
.
Do rozwiązywania równań powyższej postaci używa się metod iteracyjnych,
takich jak metoda iteracji prostej Banacha:
 ,
,czy też metoda Newtona

gdzie  jest macierzą pochodnej (zwanej także jakobianem) odwzorowania F w
punkcie xn. Czytelnika zainteresowanego, jakie założenia
na funkcję F oraz przybliżenie początkowe x0 gwarantują
zbieżność powyższych procesów iteracyjnych, odsyłamy do podręczników analizy
numerycznej [6], [18],
oraz licznych monografii, np. [15],
[11].
jest macierzą pochodnej (zwanej także jakobianem) odwzorowania F w
punkcie xn. Czytelnika zainteresowanego, jakie założenia
na funkcję F oraz przybliżenie początkowe x0 gwarantują
zbieżność powyższych procesów iteracyjnych, odsyłamy do podręczników analizy
numerycznej [6], [18],
oraz licznych monografii, np. [15],
[11].
Zauważmy, że w przypadku iteracji Newtona nie ma
potrzeby (ani sensu) odwracania macierzy F'(xn)
dla obliczenia xn+1. Kolejne przybliżenie rozwiązania
x* uzyskamy najpierw wyznaczając poprawkę na xn  ,
rozwiązując względem
,
rozwiązując względem  układ
z macierzą jakobianu, a następnie kładąc
układ
z macierzą jakobianu, a następnie kładąc  .
Oczywiście, ponieważ macierz pochodnej zmienia się w każdym kroku, rozwiązanie
jednego układu liniowego jest dużo tańsze niż najpierw wyznaczenie macierzy
odwrotnej, a dopiero potem pomnożenie jej przez wektor F(xn).
.
Oczywiście, ponieważ macierz pochodnej zmienia się w każdym kroku, rozwiązanie
jednego układu liniowego jest dużo tańsze niż najpierw wyznaczenie macierzy
odwrotnej, a dopiero potem pomnożenie jej przez wektor F(xn).
Reasumując, aby znaleźć nowe przybliżenie, musimy skonstruować macierz pochodnej, a następnie rozwiązać układ liniowy z tą macierzą. Jeśli do rozwiązywania tego układu użyjemy metody iteracyjnej (co może być uzasadnione w przypadku, gdy mamy do czynienia z macierzą rzadką dużego rozmiaru), taka metoda będzie wtedy tzw. niedokładną (ang. inexact) metodą Newtona. Możliwe są też inne modyfikacje metody Newtona, o mniejszym koszcie jednego kroku (często właśnie konstrukcja macierzy jakobianu jest zaskakująco kosztownym etapem metody Newtona). W takich przypadkach czasem stosuje się wariant, w którym macierz jakobianu jest uaktualniana dopiero po wykonaniu kilku kroków metody. Innym rozwiązaniem jest aproksymacja macierzy jakobianu przy pomocy różnic skończonych (wielowymiarowy analog metody siecznych). Zainteresowanych przeglądem rozmaitych metod rozwiązywania układów równań nieliniowych odsyłamy do zwięzłej książki [11] oraz do monografii [15].
Solvery nieliniowe dostępne w PETSc dotyczą wyłącznie metod typu Newtona. Nic dziwnego: implementacja metody iteracji (nomen omen) prostej jest co prawda nieskomplikowana, ale jej szybkość zbieżności pozostawia dużo do życzenia. Natomiast metoda Newtona jest zbieżna kwadratowo, a ponieważ w każdym kroku algorytmu musimy rozwiązać układ równań liniowych, będziemy mieli doskonałą okazję wykorzystania pełnej mocy i elastyczności SLESu.
Na niekorzyść algorytmu Newtona zdaje się działać
fakt, że wymaga on dobrego przybliżenia początkowego. Na szczęście, tę
wadę metody Newtona często można obejść w przypadku dyskretyzacji równań
różniczkowych cząstkowych, gdy "dobre'' przybliżenie początkowe można zgadnąć,
lub wyliczyć w mniej lub bardziej wyrafinowany sposób. Ponadto, w PETSc
zastosowano heurystyczne techniki "globalizujące'' zbieżność metody Newtona.
Bazują one na spostrzeżeniu, że w przypadku, gdy startujemy ze złego przybliżenia,
poprawka  jest co prawda
wektorem skierowanym w dobrym kierunku (tzn. w stronę rozwiązania x*),
ale za dużej długości. Taka sytuacja prowadzi do "przestrzelenia'' rozwiązania
i w efekcie do niezbieżności całego procesu. Dlatego wprowadza się dodatkową
operację poszukiwania optymalnej długości wektora poprawki (tzw. line
search methods). Więcej informacji na ten temat znajduje się w [11],
rozdział 8.
jest co prawda
wektorem skierowanym w dobrym kierunku (tzn. w stronę rozwiązania x*),
ale za dużej długości. Taka sytuacja prowadzi do "przestrzelenia'' rozwiązania
i w efekcie do niezbieżności całego procesu. Dlatego wprowadza się dodatkową
operację poszukiwania optymalnej długości wektora poprawki (tzw. line
search methods). Więcej informacji na ten temat znajduje się w [11],
rozdział 8.
Następnie, powołamy do życia maszynkę do rozwiązywania równań nieliniowych (czyli SNES) i przekażemy do niej dane zadania - powiemy jej, co ma być dla niej funkcją F i pochodną F' (po prostu przekażemy jej parametry - analogicznie jak w SLESie, gdzie musieliśmy przekazać SLESowi macierz układu i wektor prawej strony). W końcu, ustawimy dodatkowe parametry SNESu (albo i nie, pamiętając, że wartości domyślne są zazwyczaj rozsądnie dobrane), wygenerujemy przybliżenie początkowe i każemy SNESowi rozwiązać nasze równanie.
Tak więc, schemat wykorzystania SNESu do rozwiązania równania nieliniowego jest następujący:

Naturalnie, rozwiązaniem są pary (x,y) postaci  Rozwiązanie polega na znalezieniu zera funkcji
Rozwiązanie polega na znalezieniu zera funkcji

Jej pochodna jest oczywiście zadana macierzą następującej postaci:

Postępując zgodnie ze schematem, w naszym programie najpierw powinniśmy zdefiniować funkcję F oraz macierz pochodnej. Dla większej elegancji, kody źródłowe funkcji i jej pochodnej będziemy trzymać w osobnych plikach. Umówmy się więc, że plik uklad2x2.c będzie zawierać kod źródłowy programu głównego, tzn. funkcję main(), podczas gdy definicja procedury obliczającej F(X) znajdzie się w pliku fun2x2.c, a kod dla jakobianu F´(X) w pliku jac2x2.c. W kolejnych rozdziałach przyjrzymy się dokładnie zawartości tych plików.
fun2x2.c
#include "snes.h" /* plik z definicjami funkcji i obiektow SNES;
zawiera także dyrektywę #include "sles.h" */
/******************************************************
Procedura obliczajaca wartosci funkcji F
******************************************************/
int Funkcja(SNES MojSnes,Vec X,Vec F,void *Ctx)
{
int ierr;
Scalar *TablicaX, *TablicaF;
/* Pobierz wskazniki do tablicy z wspolrzednymi X i F */
ierr = VecGetArray(X, &TablicaX); CHKERRQ(ierr);
ierr = VecGetArray(F, &TablicaF); CHKERRQ(ierr);
/* Oblicz funkcje F */
TablicaF[0] = TablicaX[0]*TablicaX[0] + TablicaX[1]*TablicaX[1] 1.0;
TablicaF[1] = TablicaX[0] + TablicaX[1];
/* Zamknij dostep do wartosci wektorow */
ierr = VecRestoreArray(X, &TablicaX); CHKERRQ(ierr);
ierr = VecRestoreArray(F, &TablicaF); CHKERRQ(ierr);
return(0);
}
Sama definicja Funkcji() nie jest skomplikowana. Mając dany wektor X (o dwóch współrzędnych), najpierw przygotujemy się do pobrania z niego kolejnych współrzędnych:
ierr = VecGetArray(X, &TablicaX); CHKERRQ(ierr);
TablicaF[0] = TablicaX[0]*TablicaX[0] + TablicaX[1]*TablicaX[1] 1.0; TablicaF[1] = TablicaX[0] + TablicaX[1];implementując wzór

Na zakończenie musimy pamiętać, aby odblokować wektory X i F komendą VecRestoreArray(). Ta funkcja uaktualnia wektor po dokonanym dostępie do tablicy jego wartości .
jac2x2.c
/******************************************************
Definicja pochodnej F
******************************************************/
#include "snes.h"
int Jakobian(SNES MojSnes, Vec X, Mat *Jac,
Mat *Precond, MatStructure *flag, void *Ctx)
{
Scalar *x; /* Wskaznik do wartosci wektora X */
Scalar A[4]; /* Tu wpiszemy elementy macierzy Jacobiego */
int ierr;
int idx[2] = {0,1};
ierr = VecGetArray(X, &x); CHKERRQ(ierr);
/* Oblicz elementy jakobianu i wstaw do macierzy Jakobian */
A[0] = 2.0*x[0]; A[1] = 2.0*x[1]; /* pierwszy wiersz */
A[2] = 1.0; A[3] = 1.0; /* drugi wiersz */
ierr = MatSetValues(*Jac,2,idx,2,idx,A,INSERT_VALUES); CHKERRQ(ierr);
*flag = SAME_NONZERO_PATTERN;
/* Odblokuj i uaktualnij X */
ierr = VecRestoreArray(X,&x); CHKERRQ(ierr);
/* Zloz macierz */
ierr = MatAssemblyBegin(*Jac, MAT_FINAL_ASSEMBLY); CHKERRQ(ierr);
ierr = MatAssemblyEnd(*Jac, MAT_FINAL_ASSEMBLY); CHKERRQ(ierr);
return(0);
}
int Jakobian(SNES MojSnes, Vec X, Mat *Jac, Mat *Precond, MatStructure *flag, void *Ctx);
gdzie MojSnes jest obiektem SNES do którego zostanie później przekazana ta procedura, a X jest punktem w którym liczymy jakobian. Do macierzy Jac (zwróćmy uwagę, że argumentem jest wskaźnik, a nie sama macierz!) jest wpisywana wyznaczona macierz pochodnej i to jest podstawowe zadanie tej procedury: dostarczenie sposobu obliczania macierzy F'(X).
Macierz Precond oznacza macierz preconditionera (zazwyczaj będzie to ta sama macierz Jac). Parametr flag jest informacją dla PETSc, czy w następnej iteracji Newtona będzie można wykorzystać struktury preconditionera, zbudowane na użytek poprzedniej - podobnie jak w przypadku funkcji SLESSetOperators(). Wartości, jakie można przypisywać parametrowi flag były opisane w poprzedniej części (patrz Rozdział 4 na stronie 22). Ostatni parametr, Ctx, to kontekst definiowany przez użytkownika. Może on zostać wykorzystany do przekazania dodatkowych parametrów z programu potrzebnych do wyliczenia F', podobnie jak w przypadku implementacji funkcji F w procedurze Funkcja(). W naszym przykładzie nie korzystamy z Ctx.
uklad2x2.c
#include "snes.h"
#include <stdio.h>
static char help[] = "Rozwiazuje nieliniowy uklad 2x2\n\
Opcje: -x0 [Scalar] -y0 [Scalar] wartosci wspolrzednych przybl. poczatk.\n\n";
/* Deklaracje procedur obliczajacych funkcje i jakobian */
int Jakobian(SNES,Vec,Mat*,Mat*,MatStructure*,void*);
int Funkcja(SNES,Vec,Vec,void*);
/******************************************************
Program glowny
******************************************************/
int main( int argc, char **argv )
{
SNES MojSnes; /* obiekt SNES, ktorym rozwiazemy zadanie */
Vec X, R; /* wektor rozwiazania i residualny */
Mat MatJ; /* macierz jakobianu */
int ierr, flg, its;
int idx[2] = {0,1};
Scalar X0[2]; /* wspolrz. przyblizenia poczatkowego */
PetscInitialize( &argc, &argv,PETSC_NULL,help );
/* Tworzenie potrzebnych obiektow: SNESu, wektorow, macierzy jakobianu */
ierr = SNESCreate(MPI_COMM_WORLD,SNES_NONLINEAR_EQUATIONS,&MojSnes);
CHKERRA(ierr);
ierr = VecCreate(MPI_COMM_WORLD,PETSC_DECIDE,2,&X); CHKERRA(ierr);
ierr = VecDuplicate(X,&R); CHKERRA(ierr);
ierr = MatCreate(MPI_COMM_WORLD,2,2,&MatJ); CHKERRA(ierr);
/* Przekaz SNESowi funkcje i wektor residualny */
ierr = SNESSetFunction(MojSnes, R, Funkcja, PETSC_NULL); CHKERRA(ierr);
/* Przekaz SNESowi macierz jakobianu i funkcje obliczajaca jakobian */
ierr = SNESSetJacobian(MojSnes,MatJ,MatJ,Jakobian,PETSC_NULL);CHKERRA(ierr);
/* Uwzglednij opcje uruchomienia SNESu typu -snes_view -snes_monitor */
ierr = SNESSetFromOptions(MojSnes); CHKERRA(ierr);
/* Przyblizenie poczatkowe */
ierr = OptionsGetScalar( PETSC_NULL, "-x0", &X0[0], &flg ); CHKERRA(ierr);
if( !flg ) X0[0] = 1.0;
ierr = OptionsGetScalar( PETSC_NULL, "-y0", &X0[1], &flg ); CHKERRA(ierr);
if( !flg ) X0[1] = 2.0;
ierr = VecSetValues(X, 2, idx, X0, INSERT_VALUES ); CHKERRA(ierr);
ierr = VecAssemblyBegin(X); CHKERRA(ierr);
ierr = VecAssemblyEnd(X); CHKERRA(ierr);
/* Rozwiaz! */
ierr = SNESSolve(MojSnes,X,&its); CHKERRA(ierr);
PetscPrintf(MPI_COMM_WORLD, "Liczba iteracji = %d\n", its);
PetscPrintf(MPI_COMM_WORLD,"Wektor rozwiazania:\n");
ierr = VecView(X, VIEWER_STDOUT_WORLD); CHKERRA(ierr);
/* Zwolnij pamiec */
ierr = VecDestroy(X); CHKERRA(ierr);
ierr = VecDestroy(R); CHKERRA(ierr);
ierr = MatDestroy(MatJ); CHKERRA(ierr);
ierr = SNESDestroy(MojSnes); CHKERRA(ierr);
/* Koniec bajki */
PetscFinalize();
return 0;
}
Oto przykładowy wynik uruchomienia powyższego programu:
11:/home/staff/przykry/Petsc> test Liczba iteracji = 7 Wektor rozwiazania: -0.707107 0.707107
 = 0.7071...).
Zobaczmy teraz, jakie są szczegóły poszczególnych kroków algorytmu. Na
początku pliku uklad2x2.c pojawiają się dyrektywy:
= 0.7071...).
Zobaczmy teraz, jakie są szczegóły poszczególnych kroków algorytmu. Na
początku pliku uklad2x2.c pojawiają się dyrektywy:#include "snes.h" #include <stdio.h>
static char help[] = "Rozwiazuje nieliniowy uklad 2x2\n\ Opcje: -x0 [Scalar] -y0 [Scalar] wartosci wspolrzednych przybl. poczatk.\n\n";
int Jakobian(SNES,Vec,Mat*,Mat*,MatStructure*,void*); int Funkcja(SNES,Vec,Vec,void*);
Na początku funkcji main() deklarujemy kilka zmiennych roboczych:
SNES MojSnes; /* obiekt SNES, ktorym rozwiazemy zadanie */
Vec X, R; /* wektor rozwiazania i residualny */
Mat MatJ; /* macierz jakobianu */
int ierr, flg, its;
int idx[2] = {0,1};
Scalar X0[2]; /* wspolrz. przyblizenia poczatkowego */
PetscInitialize( &argc, &argv,PETSC_NULL,help );
SNESCreate(MPI_COMM_WORLD,SNES_NONLINEAR_EQUATIONS,&MojSnes);
VecCreate(MPI_COMM_WORLD,PETSC_DECIDE,2,&X); VecDuplicate(X,&R);
MatCreate(MPI_COMM_WORLD,2,2,&MatJ);
Druga funkcja przekazuje do SNESu macierz jakobianu i metodę uaktualniania go: int SNESSetJacobian( SNES MojSnes, Mat Jac, Mat Prec, int (*Jakobian)(SNES, Vec, Mat*, Mat*, MatStructure*,void*), void *Ctx ) Podobnie jak poprzednio, pierwszym argumentem jest MojSnes - obiekt SNESu, do którego chcemy przekazać funkcję obliczającą F'(X), tzn. macierz jakobianu. Drugim argumentem jest więc macierz jakobianu Jac, do której będą wprowadzane wartości elementów, obliczone w procedurze Jakobian(). Trzecim argumentem jest macierz preconditionera Prec dla macierzy Jac, przydatna przy rozwiązywaniu układów z tą macierzą. Zazwyczaj wystarcza podać jako Prec po prostu tą samą macierz Jac. Kolejny, czwarty argument to nazwa procedury - u nas: Jakobian() - obliczającej elementy macierzy jakobianu F'(X). Jak widać z powyższego formatu, SNES (i kompilator...) oczekuje, że funkcja Jakobian()jest deklarowana jako int Jakobian(SNES MojSnes,Mat *Jac,Mat *Prec,int *flg,void *Ctx) i właśnie dlatego w taki, a nie inny sposób definiowaliśmy naszą procedurę Jakobian() w pliku jac2x2.c powyżej. Ostatni argument dla SNESSetJacobian() to wskaźnik do kontekstu, wykorzystywanego przez procedurę Jakobian() i zawierającego dane używane przy wyznaczaniu macierzy F'(X).
Ponieważ nasze procedury: Funkcja()oraz Jakobian() nie korzystają z kontekstu użytkownika, jako ostatni argument wywołania SNESSetFunction() i SNESSetJacobian() podajemy PETSC_NULL:
/* Przekaz SNESowi funkcje i wektor residualny */ SNESSetFunction(MojSnes, R, Funkcja, PETSC_NULL); /* Przekaz SNESowi macierz jakobianu i funkcje obliczajaca jakobian */ SNESSetJacobian(MojSnes,MatJ,MatJ,Jakobian,PETSC_NULL); /* Uwzglednij opcje uruchomienia SNESu typu -snes_view -snes_monitor */ SNESSetFromOptions(MojSnes);
Wszystkie dane o równaniu są już przekazane do SNESu, opcje ustawione, moglibyśmy więc już rozwiązać równanie... jeśli tylko określilibyśmy przybliżenie początkowe. Przybliżenie początkowe (analogicznie jak w SLESie) wpiszemy wprost do wektora rozwiązania. Właśnie do tego przydadzą się nam tablice X0 i idx, zadeklarowane na początku programu.
/* Przyblizenie poczatkowe */ OptionsGetScalar( PETSC_NULL, "-x0", &X0[0], &flg ); if( !flg ) X0[0] = 1.0; OptionsGetScalar( PETSC_NULL, "-y0", &X0[1], &flg ); if( !flg ) X0[1] = 2.0; VecSetValues(X, 2, idx, X0, INSERT_VALUES ); ierr = VecAssemblyBegin(X); CHKERRA(ierr); ierr = VecAssemblyEnd(X); CHKERRA(ierr);
18:/home/staff/przykry/Petsc> test -y0 5.0
Po określeniu przybliżenia początkowego nie pozostaje nam już nic innego, jak nakazać SNESowi rozwiązanie naszego zadania:
SNESSolve(MojSnes,X,&its);
PetscPrintf(MPI_COMM_WORLD, "Liczba iteracji = %d\n", its); PetscPrintf(MPI_COMM_WORLD,"Wektor rozwiazania:\n"); VecView(X, VIEWER_STDOUT_WORLD);
VecDestroy(X); VecDestroy(R); MatDestroy(MatJ); SNESDestroy(MojSnes); PetscFinalize();
Makefile include $(PETSC_DIR)/bmake/$(PETSC_ARCH)/base CC = gcc -DPETSC_LOG CLINKER = gcc CFLAGS = $(PCONF) $(PETSC_INCLUDE) $(BS_INCLUDE) LIBBASE = libpetscsles libpetscsnes OPTS = -O BOPT = O OBJECTS_EX1 = uklad2x2.o fun2x2.o jac2x2.o EXECUTABLE = test ex1: $(OBJECTS_EX1) -$(CLINKER) -o $(EXECUTABLE) $(OBJECTS_EX1) \ -lm $(PETSC_LIB) # # ex1 source files # uklad2x2.o: uklad2x2.c $(CC) $(CFLAGS) $(OPTS) -c uklad2x2.c fun2x2.o: fun2x2.c $(CC) $(CFLAGS) $(OPTS) -c fun2x2.c jac2x2.o: jac2x2.c $(CC) $(CFLAGS) $(OPTS) -c jac2x2.c


Dla prostoty, to równanie będziemy aproksymować metodą różnic skończonych, dostając problem dyskretny postaci:
 znaleźć
znaleźć  spełniający
spełniający
 dla
dla  ,
, .
.Jak widzimy, mamy tym razem do czynienia z układem N równań nieliniowych, gdzie N oznacza liczbę wewnętrznych węzłów siatki (warunki brzegowe Dirichleta wyznaczają od razu u0 i uN+1, więc możemy ograniczyć się jedynie do obliczenia u w pozostałych węzłach). Jeśli więc siatka jest gęsta, to rozmiar zadania nieliniowego jest bardzo duży, a macierz pochodnej rozrzedzona. Właśnie to powoduje, że stosowanie PETSc do problemów pochodzących z dyskretyzacji nieliniowych równań różniczkowych cząstkowych zaczyna być sensowne. Narzędzia dostępne w PETSc (a więc także w SNESie) były tworzone właśnie z myślą o efektywnych metodach rozwiązywania problemów powstałych z dyskretyzacji równań różniczkowych cząstkowych.
Zacznijmy od przeanalizowania problemu. To jest
bardzo ważne, albowiem od tego, w jaki sposób podejdziemy do zagadnienia,
będzie zależało wiele naszych następnych posunięć. Mamy do czynienia z
nieliniowym zagadnieniem dyskretnym, które zamierzamy rozwiązywać metodą
Newtona. Musimy więc najpierw przeformułować to zagadnienie jako zero pewnego
odwzorowania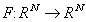 . Oznaczając przez
uh wektor rozwiązania:
. Oznaczając przez
uh wektor rozwiązania:
 ,
,możemy zapisać zagadnienie dyskretne w postaci wektorowej: 
 ,
,gdzie Ah oznacza macierz (minus) laplasjanu, a Bh macierz dyskretyzacji pierwszej pochodnej różnicą centralną, tzn.:

Zwróćmy uwagę, że tym razem operację mnożenia wektorów  rozumiemy nie w sensie iloczynu skalarnego, tylko jako operację mnożenia
odpowiadających współrzędnych.
rozumiemy nie w sensie iloczynu skalarnego, tylko jako operację mnożenia
odpowiadających współrzędnych.
Przy takim zapisie łatwo już określić funkcję, której zerem będzie rozwiązanie naszego zagadnienia:

Jej pochodną będzie naturalnie taki operator liniowy  ,
który w działaniu na wektor w da wektor F'(u)w, określony
przez
,
który w działaniu na wektor w da wektor F'(u)w, określony
przez 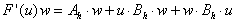 . Określiliśmy
więc
. Określiliśmy
więc  oraz wyprowadziliśmy
wzór na F'. Jak pamiętamy, SNES będzie potrzebował procedur obliczających
oraz wyprowadziliśmy
wzór na F'. Jak pamiętamy, SNES będzie potrzebował procedur obliczających  i
i .
Musimy teraz zastanowić się, jak zaimplementować te funkcje w PETSc. Tutaj
pojawia się następna zaleta zapisu wektorowego: daje się on bardzo łatwo
"przetłumaczyć" na kod źródłowy PETSc!
.
Musimy teraz zastanowić się, jak zaimplementować te funkcje w PETSc. Tutaj
pojawia się następna zaleta zapisu wektorowego: daje się on bardzo łatwo
"przetłumaczyć" na kod źródłowy PETSc!
   |
 |
macierz laplasjanu pomnożona przez X funkcją MatMult() |
 |
 |
wynik mnożenia macierzy pierwszej pochodnej przez X, razy (po współrzędnych) X - funkcje MatMult() oraz VecPointwiseMult(), |
 |
 |
prawa strona |
Czy z pochodną F'(u) pójdzie nam równie łatwo?...
 możemy reprezentować jako macierz
możemy reprezentować jako macierz  .
Zwróćmy jednak uwagę na pewien detal: było nam wygodniej zapisać pochodną
F nie jako macierz pochodnych cząstkowych, lecz jako operator, którego
wartości na zadanym wektorze w są zdefiniowane pewnym wzorem. W
taki właśnie sposób definiuje się w PETSc macierze powłokowe (shell
matrices), o których była mowa na stronie 18. Idea macierzy powłokowych
wyjątkowo dobrze sprawdzi się w naszym zagadnieniu:
.
Zwróćmy jednak uwagę na pewien detal: było nam wygodniej zapisać pochodną
F nie jako macierz pochodnych cząstkowych, lecz jako operator, którego
wartości na zadanym wektorze w są zdefiniowane pewnym wzorem. W
taki właśnie sposób definiuje się w PETSc macierze powłokowe (shell
matrices), o których była mowa na stronie 18. Idea macierzy powłokowych
wyjątkowo dobrze sprawdzi się w naszym zagadnieniu:Ktoś bardzo początkujący mógłby pomyśleć o takiej realizacji:
int Funkcja( SNES MojSnes, Vec X, Vec F, void * Ctx )
{
Mat A, B;
/* generuj A i B, a nastepnie przypisz im wartosci */
MatCreate(A); MatCreate(B);
MatSetValues(A); ...itd....
/* uzyj A i B do obliczenia F */
MatMult(A); VecAXPY(); MatMult(B); itd...
/* zwolnij macierze A i B */
MatDestroy(A); MatDestroy(B);
}
Podstawową wadą zaproponowanej procedury jest wielokrotne robienie tego samego: generowanie wciąż tych samych macierzy. Najprostszym sposobem usunięcia tego problemu byłoby jednokrotne utworzenie i wygenerowanie elementów macierzy A i B (raz, a dobrze!) gdzieś na zewnątrz procedury Funkcja(). Świetnie, ale jak skorzystać z tych macierzy wewnątrz procedury? Znów, najszybciej przychodzi nam do głowy rozwiązanie najprostsze, ale jednocześnie najbardziej bałaganiarskie: po prostu, trzeba zadeklarować te macierze jako zmienne globalne!
Mat A, B; /* nasze macierze jako zmienne globalne:
deklarowane na zewnatrz wszystkich funkcji */
/******************************************************
Program glowny
******************************************************/
int main(int argc, char **argv)
{
/* generuj A i B, a nastepnie przypisz im wartosci */
MatCreate(A); MatCreate(B);
MatSetValues(A); ...itd...
/* ......... */
SNESSetFunction( Funkcja );
/* ......... */
/* zwolnij macierze A i B */
MatDestroy(A); MatDestroy(B);
return(0);
}
/******************************************************
Procedura obliczajaca F(X)
******************************************************/
int Funkcja( SNES MojSnes, Vec X, Vec F, void * Ctx )
{
/* uzyj A i B do obliczenia F */
MatMult(A); VecAXPY(); MatMult(B); itd...
return(0);
}
To już jest całkiem dobre, a zwłaszcza - znacznie bardziej efektywne, niż
poprzednia propozycja. Niemniej, kiedy wiemy, jak to zrobić efektywnie,
powinniśmy spróbować zrobić to elegancko i temu właśnie będzie służył tytułowy
koncept kontekstu użytkownika (pomysł na grę słów zaczerpnęliśmy
od B.Smitha). Cóż, eleganckim rozwiązaniem byłoby przekazanie do Funkcji()macierzy
Ah i Bh (uprzednio wygenerowanych na
zewnątrz procedury mnożenia!) w postaci parametrów. Z tym jednak
pozornie jest kłopot: format Funkcji() jest sztywny i nie możemy dowolnie
modyfikować listy jej argumentów. Na szczęście, ostatnim dopuszczalnym
argumentem funkcji jest wskaźnik do kontekstu użytkownika, zawierającego
pomocnicze dla procedury obiekty i właśnie w ten sposób przekażemy nasze
macierze!
Jak taki kontekst powinien wyglądać? Zasadniczo, będzie to struktura w C, zawierająca parametry, które zechcemy przekazać do Funkcji(). Tymi parametrami mogą być macierze, wektory robocze (o tak, przydadzą się!), a nawet wskaźniki do funkcji udostępniających zmienne lokalne funkcjom używającym naszego kontekstu. Taka struktura, ze zmiennymi lokalnymi, funkcjami dostępu itp. mocno pachnie porządną klasą w C++ i tak jest w rzeczywistości: programowanie z użyciem kontekstów ma wiele cech programowania obiektowego.
Wracając do naszej konkretnej sytuacji, kontekst, jaki chcielibyśmy przekazać do Funkcji(), powinien zawierać dwie macierze: dyskretyzacji laplasjanu i pierwszej pochodnej. Kontekst ma być strukturą, więc wygodnie nam będzie najpierw zdefiniować odpowiedni typ strukturalny:
typedef struct {
Mat A;
Mat B;
} USERCTX;
int main(int argc, char **argv)
{
USERCTX UserCtx;
/* inicjalizacja kontekstu */
USERCTXCreate( &UserCtx );
/* ....wykorzystanie kontekstu.... */
SNESSetFunction( MojSnes, X, R, Funkcja, (void *) &UserCtx );
/* zwolnienie kontekstu */
USERCTXDestroy( &UserCtx );
}
int USERCTXCreate( USERCTX *UserCtx )
{
MatCreate( &UserCtx->A ); MatCreate( &UserCtx->B );
MatSetValues( UserCtx->A );
...itd....
}
int USERCTXDestroy( USERCTX *UserCtx )
{
MatDestroy( UserCtx->A ); ...itd...
}
int Funkcja( SNES MojSnes, Vec X, Vec F, void * Ctx )
{
USERCTX *UserCtx;
/* najpierw rzutujemy wskaznik na odpowiedni typ */
UserCtx = (USERCTX *) Ctx;
/* uzyj A i B do obliczenia F */
MatMult( UserCtx->A, X, F ); VecAXPY(); MatMult(); itd...
return(0);
}
user.h
static Scalar zero = 0.0;
static Scalar one = 1.0;
static Scalar mone = -1.0;
typedef struct
{
Mat Lap; /* macierz laplasjanu */
Mat Poch; /* macierz pierwszej pochodnej */
Vec X; /* wektor rozwiazania */
Vec F; /* prawej strony */
Vec R; /* residuum */
Vec y; /* pomocniczy */
Vec z; /* pomocniczy */
} USERCTX;
Dodatkowo, zdefiniowaliśmy parę stałych typu Scalar, bardzo przydatnych do wykonania operacji obliczenia sumy lub różnicy dwóch wektorów przy pomocy funkcji VecAXPY(). Będą one dostępne w każdym pliku zawierającym dyrektywę #include "user.h".
cdfun.c
#include "snes.h" /* plik z definicjami funkcji i obiektow SNES; zawiera takze
dyrektywe #include "sles.h" */
#include "user.h" /* kontekst uzytkownika i przydatne stale */
/******************************************************
Procedura obliczajaca wartosci funkcji F
******************************************************/
int Funkcja(SNES MojSnes,Vec X,Vec F,void *Ctx)
{
int ierr;
USERCTX *UserCtx = (USERCTX *)Ctx;
/* Pomnoz przez pochodna centralna... */
ierr = MatMult( UserCtx->Poch, X, UserCtx->y ); CHKERRQ(ierr);
/* ...i domnoz przez u */
ierr = VecPointwiseMult( X, UserCtx->y, F ); CHKERRQ(ierr);
/* Pomnoz przez laplasjan */
ierr = MatMult( UserCtx->Lap, X, UserCtx->y ); CHKERRQ(ierr);
/* Zsumuj! */
ierr = VecAXPY( &one, UserCtx->y, F ); CHKERRQ(ierr);
ierr = VecAXPY( &mone, UserCtx->F, F ); CHKERRQ(ierr);
return(ierr);
}
cdjac.c
/******************************************************
Definicja pochodnej F
******************************************************/
#include "snes.h"
#include "user.h"
/* Implementacja funkcji Jakobian(), przekazywanej do SNESu */
int Jakobian(SNES MojSnes, Vec X, Mat *Jac,
Mat *Precond, MatStructure *flag, void *Ctx)
{
*flag = SAME_NONZERO_PATTERN;
return(0);
}
/* Implementacja mnozenia przez macierz jakobianu */
int MultJakobian( Mat Jac, Vec VecIn, Vec VecOut )
{
int ierr;
USERCTX *UserCtx;
/* Najpierw musimy wydobyc kontekst */
ierr = MatShellGetContext( Jac, (void **)&UserCtx );CHKERRQ(ierr);
/* Pomnoz przez pochodna centralna... */
ierr = MatMult( UserCtx->Poch, VecIn, UserCtx->y ); CHKERRQ(ierr);
/* ...i domnoz przez u */
ierr = VecPointwiseMult( UserCtx->X, UserCtx->y, VecOut ); CHKERRQ(ierr);
/* Pomnoz przez laplasjan */
ierr = MatMult( UserCtx->Lap, VecIn, UserCtx->y ); CHKERRQ(ierr);
/* Zsumuj! */
ierr = VecAXPY( &one, UserCtx->y, VecOut ); CHKERRQ(ierr);
ierr = MatMult( UserCtx->Poch, UserCtx->X, UserCtx->y ); CHKERRQ(ierr);
ierr = VecPointwiseMult( VecIn, UserCtx->y, UserCtx->z ); CHKERRQ(ierr);
ierr = VecAXPY( &one, UserCtx->z, VecOut ); CHKERRQ(ierr);
return(ierr);
}
Ponieważ wszystko załatwia operacja mnożenia, jest to dobre miejsce na zdefiniowanie procedury mnożenia wektora przez macierz jakobianu. Ta procedura zostanie potem przekazana jako argument MatShellSetOperation(), dopełniając definiowanie powłokowej macierzy F'(X). Procedurę MultJakobian(), definiującą de facto macierz F'(X), wykorzystamy przy okazji tworzenia powłokowej macierzy jakobianu w programie głównym.
cd.c
#include <stdio.h>
#include <math.h> /* zawiera funkcje sin(x) */
#include "snes.h"
#include "user.h"
static char help[] = "Rozwiazuje nieliniowe jednowymiarowe rownanie\n\
konwekcji-dyfuzji na odcinku [0,L]\n\
Opcje: -N [int] liczba wewnetrznych wezlow siatki\n\
-L [Scalar] dlugosc odcinka\n\n";
/* Deklaracje procedur obliczajacych funkcje i jakobian */
extern int MultJakobian( Mat, Vec, Vec);
int Jakobian(SNES,Vec,Mat*,Mat*,MatStructure*,void*);
int Funkcja(SNES,Vec,Vec,void*);
int USERCTXCreate(USERCTX*,int,Scalar);
int USERCTXDestroy(USERCTX*);
/**************************************************************
Program glowny
**************************************************************/
int main( int argc, char **argv )
{
SNES MojSnes; /* obiekt SNES, ktorym rozwiazemy zadanie */
Mat MatJ; /* macierz jakobianu */
USERCTX UserCtx; /* kontekst uzytkownika */
int n; /* liczba wezlow siatki */
static Scalar L = M_PI; /* dlugosc przedzialu calkowania,
zatem krok siatki h = L/n */
int ierr, flg, its;
PetscInitialize( &argc, &argv,PETSC_NULL,help );
/* Interpretacja opcji uzytkownika */
ierr = OptionsGetInt( PETSC_NULL, "-N", &n, &flg ); CHKERRA(ierr);
if( !flg ) n = 3;
ierr = OptionsGetScalar( PETSC_NULL, "-L", &L, &flg );
CHKERRA(ierr);
/* Tworzenie potrzebnych obiektow: SNESu, wektorow, macierzy pomocniczych */
ierr = SNESCreate(MPI_COMM_WORLD,SNES_NONLINEAR_EQUATIONS,&MojSnes); CHKERRA(ierr);
ierr = USERCTXCreate( &UserCtx, n, L ); CHKERRA(ierr);
VecView( UserCtx.F, VIEWER_DRAWX_WORLD ); PetscSleep(5);
/* Przekaz SNESowi funkcje i wektor residualny */
ierr = SNESSetFunction(MojSnes, UserCtx.R, Funkcja, &UserCtx); CHKERRA(ierr);
/* Przekaz SNESowi macierz jakobianu i funkcje obliczajaca jakobian */
ierr = MatCreateShell(MPI_COMM_WORLD,n,n,n,n,&UserCtx,&MatJ); CHKERRA(ierr);
ierr = MatShellSetOperation(MatJ, MATOP_MULT, MultJakobian ); CHKERRA(ierr);
ierr = SNESSetJacobian(MojSnes,MatJ,MatJ,Jakobian,&UserCtx); CHKERRA(ierr);
/* Uwzglednij opcje uruchomienia SNESu typu -snes_view -snes_monitor */
ierr = SNESSetFromOptions(MojSnes); CHKERRA(ierr);
/* Rozwiaz! */
ierr = SNESSolve(MojSnes,UserCtx.X,&its); CHKERRA(ierr);
PetscPrintf(MPI_COMM_WORLD, "Liczba iteracji = %d\n", its);
PetscPrintf(MPI_COMM_WORLD,"Wektor rozwiazania:\n");
ierr = VecView(UserCtx.X, VIEWER_DRAWX_WORLD); CHKERRA(ierr);
PetscSleep( 10 );
/* Zwolnij pamiec */
ierr = USERCTXDestroy(&UserCtx); CHKERRA(ierr);
ierr = MatDestroy(MatJ); CHKERRA(ierr);
ierr = SNESDestroy(MojSnes); CHKERRA(ierr);
PetscFinalize();
return 0;
}
/**************************************************************
Funkcje zwiazane z kontekstem uzytkownika
**************************************************************/
int USERCTXCreate( USERCTX * UserCtx, int n, Scalar L )
{
extern int GenerujLaplasjan1D( Mat, Scalar );
extern int GenerujPochodnaCentralna1D( Mat, Scalar );
int ierr=0;
int i;
double val;
double h = L/(n+1); /* krok siatki */
/* tworzymy wektory uzywane w kontekscie naszego zagadnienia */
ierr = VecCreate(MPI_COMM_WORLD,PETSC_DECIDE,n,&UserCtx->X); CHKERRQ(ierr);
ierr = VecDuplicate(UserCtx->X,&UserCtx->R); CHKERRQ(ierr);
ierr = VecDuplicate(UserCtx->X,&UserCtx->F); CHKERRQ(ierr);
ierr = VecDuplicate(UserCtx->X,&UserCtx->y); CHKERRQ(ierr);
ierr = VecDuplicate(UserCtx->X,&UserCtx->z); CHKERRQ(ierr);
/* tworzymy macierze uzywane w kontekscie naszego zagadnienia */
ierr = MatCreate(MPI_COMM_WORLD,n,n,&UserCtx->Lap); CHKERRQ(ierr);
ierr = MatCreate(MPI_COMM_WORLD,n,n,&UserCtx->Poch); CHKERRQ(ierr);
/* generujemy elementy macierzy pomocniczych */
ierr = GenerujLaplasjan1D( UserCtx->Lap, (Scalar) h ); CHKERRQ(ierr);
ierr = MatView( UserCtx->Lap, VIEWER_DRAWX_WORLD ); CHKERRQ(ierr);
PetscSleep( 3 );
ierr = GenerujPochodnaCentralna1D( UserCtx->Poch, (Scalar) h );CHKERRQ(ierr);
ierr = MatView( UserCtx->Poch, VIEWER_DRAWX_WORLD ); CHKERRQ(ierr);
PetscSleep( 3 );
/* generujemy prawa strone */
for( i = 1 ; i <= n ; i++ )
{
val = sin(i*h) + sin(i*h)*cos(i*h); /* rozwiazanie u(x) = sin(x) */
VecSetValue( UserCtx->F, i-1, (Scalar)val, INSERT_VALUES );
}
/* Przyblizenie poczatkowe */
/* Zakladamy, ze zerowe... Od razu wpisujemy do wektora rozwiazania X */
ierr = VecSet(&zero, UserCtx->X); CHKERRQ(ierr);
return(ierr);
}
int USERCTXDestroy( USERCTX * UserCtx )
{
int ierr;
ierr = VecDestroy(UserCtx->X); CHKERRQ(ierr);
ierr = VecDestroy(UserCtx->R); CHKERRQ(ierr);
ierr = VecDestroy(UserCtx->F); CHKERRQ(ierr);
ierr = VecDestroy(UserCtx->z); CHKERRQ(ierr);
ierr = VecDestroy(UserCtx->y); CHKERRQ(ierr);
ierr = MatDestroy(UserCtx->Lap); CHKERRQ(ierr);
ierr = MatDestroy(UserCtx->Poch); CHKERRQ(ierr);
return(ierr);
}
Po wstępnych #include następuje lista prototypów procedur - zdefiniowanych gdzie indziej, np. w innych plikach - a wykorzystywanych poniżej. Przydaje się to kompilatorowi dla pełniejszego sprawdzania poprawności wywołań tych funkcji. W samej funkcji main() deklarujemy na początku kilka zmiennych lokalnych, w tym obiekt SNESu MojSnes, macierz (za chwilę okaże się, że powłokową) jakobianu MatJ oraz kontekst UserCtx, zawierający m.in. macierze laplasjanu i pierwszej pochodnej, a także wektory rozwiązania i residuum. Zaczynamy klasycznie, od inicjalizacji PETSc oraz ściągnięcia opcji użytkownika:
PetscInitialize( &argc, &argv,PETSC_NULL,help ); /* Interpretacja opcji uzytkownika */ OptionsGetInt( PETSC_NULL, "-N", &n, &flg ); if( !flg ) n = 3; OptionsGetScalar( PETSC_NULL, "-L", &L, &flg );
/* Tworzenie potrzebnych obiektow: SNESu, wektorow, macierzy pomocniczych */ SNESCreate(MPI_COMM_WORLD,SNES_NONLINEAR_EQUATIONS,&MojSnes); USERCTXCreate( &UserCtx, n, L );
/* Przekaz SNESowi funkcje i wektor residualny */ SNESSetFunction(MojSnes, UserCtx.R, Funkcja, &UserCtx);
MatCreateShell(MPI_COMM_SELF,n,n,n,n,&UserCtx,&MatJ); MatShellSetOperation(MatJ, MATOP_MULT, MultJakobian );
SNESSetJacobian(MojSnes,MatJ,MatJ,Jakobian,&UserCtx);
 , z jednorodnymi
warunkami brzegowymi Dirichleta.
, z jednorodnymi
warunkami brzegowymi Dirichleta.
Wybór schematu numerycznego dla aproksymacji tego równania nie jest trywialny.
Możemy teraz wybierać stopień wielomianu interpolacyjnego
używanego do wyznaczenia lepszego czynnika skalującego: -snes_eq_ls
[quadratic,cubic], to znaczy, odpowiednio, funkcją kwadratową lub sześcienną.
Więcej szczegółów Czytelnik znajdzie u Kelley'a [11]
oraz w [14]. Dodatkowo, możliwy jest
także wariant
-snes_eq_ls basic. Taki wybór oznacza rezygnację z techniki
line search i pozostanie przy klasycznej metodzie Newtona.
Metody wyszukiwania kierunkowego wymagają ustawienia
pewnych parametrów relaksacyjnych,
-snes_eq_ls_alpha <alpha>, -snes_eq_ls_maxstep
<max>, -snes_eq_ls_steptol <tol>, gdzie oznaczenia są zgodne
z [4]. Na szczęście, predefiniowane
wartości tych parametrów są zazwyczaj rozsądne dla wielu zagadnień.
 atol,
atol, rtol,
rtol, stol.
stol.ierr = SNESSetTolerances(SNES snes,double rtol,double atol,double stol, int maxits,int maxfcts);
lub z linii komend: -snes_atol <atol>, -snes_rtol
<rtol>, -snes_stol <stol>,
-snes_max_it <maxits> oraz -snes_max_funcs <maxfcts>.
Zmianę opcji SLESu wykorzystywanego przez SNES możemy uzyskać z programu, wyłuskując kontekst SLESu funkcją int SNESGetSLES( SNES MojSnes, SLES * MojSnesSles ) a następnie używając procedur typu SLESGetKSP() lub SLESGetPC(). Naturalnie, to samo uzyskamy podając odpowiednie opcje w linii komend.

Zazwyczaj programy (w początkowym stadium) nie są wolne od błędów. Można się w szczególności obawiać, że zaprogramowana przez nas macierz jakobianu nie jest idealna. Na szczęście, możemy porównać działanie programu z naszą macierzą jakobianu i programu, w którym zostanie użyta jej aproksymacja metodą różnic skończonych. W wypadku, gdy używamy macierzy typu powłokowego, możemy naszą funkcję Jakobian(), realizującą działanie jakobianu, zastąpić jej aproksymacją generowaną wewnętrznie przez PETSc metodą różnic skończonych: wystarczy uruchomić program z opcją -snes_mf. Podobny skutek można uzyskać (ale już tylko dla niepowłokowej macierzy jakobianu) stosując opcję -snes_fd. Nie znamy bliżej różnic między tymi opcjami. Po co stosować te opcje? Na pewno przydadzą się one tym, którzy nie potrafią (lub nie mają czasu) implementować funkcji Jakobian(). Czasem macierz jakobianu może być skomplikowana! Autorzy oficjalnego manuala twierdzą ponadto, że możemy w ten sposób łatwo sprawdzić, czy poprawnie zaimplementowaliśmy jakobian: bo jeśli nie, to, rozwiązując zagadnienie raz z własnym jakobianem, a drugi raz z opcją -snes_mf, powinniśmy dostać istotnie różniące się wyniki. To prawda, ale od razu chcielibyśmy ostrzec wszystkich pesymistów: jeśli postępując w wyżej opisany sposób, rzeczywiście dostaniecie różne wyniki (rozwiązania, liczbę iteracji, itp.) nie wpadajcie w desperację. Jest bardzo prawdopodobne, że po prostu rozwiązujecie swoje zadanie z niedobrymi parametrami! Pamiętajcie, że wyznaczenie aproksymacji jakobianu jest numerycznie bardzo kosztowne (wymaga za każdym razem obliczania wielu wartości Funkcji()) a jednym z ograniczeń ustawianym w SNESie jest właśnie liczba wywołań funkcji!
Ponadto, każdy SNES posiada (oczywiście!...) swoją przeglądarkę, wyświetlającą parametry wywołania SNESu: int SNESView(SNES snes,Viewer viewer), gdzie, jak zwykle, jako viewer możemy podać VIEWER_STDOUT_WORLD. Z linii komend uzyskamy ten sam efekt podając opcję -snes_view. Oto przykładowy wynik obejrzenia SNESu:
SNES Object: method: ls line search variant: SNESCubicLineSearch alpha=0.0001, maxstep=1e+08, steptol=1e12 maximum iterations=50, maximum function evaluations=1000 tolerances: relative=1e-08, absolute=1e-50, truncation=1e-12, solution=1e-08 total number of linear solver iterations=13 total number of function evaluations=6 KSP Object: method: gmres GMRES: restart=30, using Modified GramSchmidt Orthogonalization maximum iterations=10000, initial guess is zero tolerances: relative=1e-05, absolute=1e-50, divergence=10000 left preconditioning PC Object: method: none linear system matrix = precond matrix: Matrix Object: type=MATSHELL, rows=343, cols=343
|
|
|
| int SNESCreate(MPI_Comm comm,SNESProblemType mode,SNES *MojSnes) | tworzy obiekt typu SNES; |
| int SNESSetFunction(SNES MojSnes, Vec R, int (*Funkcja)(SNES,Vec,Vec,void*),void *FunCtx) | Przekazuje SNESowi procedurę obliczającą F(X); |
| int SNESSetJacobian(SNES MojSnes,Mat Jac,Mat Prec, int(*Jakobian)(SNES,Vec,Mat*,Mat*,MatStructure*,void*), void *JacCtx) | Przekazuje SNESowi procedurę obliczającą jakobian F'(X); |
|
int SNESSetFromOptions(SNES MojSnes) |
ustawia opcje SNESu zgodnie z argumentami przekazanymi w linii komend przy wywołaniu programu; |
| int SNESSolve(SNES MojSnes,Vec X,int *its) | Rozwiązuje równanie F(X)=0; |
| int SNESView(SNES MojSnes,Viewer viewer) | Wypisuje parametry SNESu. |
int Jakobian(SNES MojSnes,Vec X,Mat *Jac,Mat *Prec,int *flag, void *JacCtx); funkcja wyznaczająca macierz jakobianu Jac i preconditionera Prec na podstawie wartości rozwiązania na kolejnym kroku X; w wyniku musi ustawiać wartość paramteru flag identyfikującego zmianę struktury macierzy Prec, analogicznie jak w SLESSetOperators(); opis użycia tej funkcji w przypadku, gdy te macierze są macierzami powłokowymi znajduje się w rozdziale "Definicja funkcji F" na stronie 57.
|
|
SNESCreate(MPI_COMM_WORLD, SNES_NONLINEAR_EQUATIONS,
&MojSnes);
SNESSetFunction(MojSnes, X, Funkcja, (void
*) &UserCtx); oraz
SNESSetJacobian(MojSnes, Jac, Jac, Jakobian, (void *) &UserCtx);
|
Zarówno procedura obliczająca funkcję F(X), jak procedura obliczająca jakobian F'(X), muszą być ściśle określonego typu. Przed użyciem SNESu, musimy wygenerować niezłe przybliżenie początkowe. Efektywność całego procesu rozwiązywania równania będzie zależała przede wszystkim od sposobu implementacji F(X) oraz F'(X). Warto także zastanowić się, czy dobrze są dobrane parametry SNESu.
Oprócz SLESu i SNESu, mamy także moduł TS (ang. time stepping), z implementacją metod różnicowych rozwiązywania wielkich układów równań różniczkowych zwyczajnych powstałych na przykład po dyskretyzacji (względem zmiennej przestrzennej) równania parabolicznego. Dodajmy, że PETSc ma możliwość wykorzystywania w tym celu także biblioteki zewnętrznej PVODE autorstwa A.Hindmarsha i in., będącej równoległą wersją biblioteki CVODE (dość wyrafinowanych metod wielokrokowych).
PETSc posiada wiele opcji profilowania kodu. Jedną
z nich można uruchomić dodając do linii komend
-petsc_log. Użytkownik ma wtedy możliwość sprawdzenia, ile
czasu CPU (lub jaki procent obliczeń) pochłania wybrany fragment kodu.
Można też śledzić sposób wykorzystywania pamięci, obciążenie poszczególnych
procesorów, itp. Poza wydrukiem tekstowym, jest także możliwość obejrzenia
przebiegu pracy programu w okienku graficznym. Przyznajemy, że trochę żałujemy,
iż do tej pory nie zaczęliśmy w pełni wykorzystywać tych opcji.
Dodatkowo, dla prawdziwych i porządnych programistów-profesjonalistów, PETSc daje możliwość podłączenia debuggera - zarówno dopiero po wystąpieniu błędu, jak też od początku pracy programu. Debugger może się podłączać na każdym procesorze w osobnym okienku - każdy, kto doświadczył koszmaru programowania równoległego, wie, jakie to może być ważne. I znów, jako nieprofesjonaliści, nie korzystamy z tej sposobności, gorzej, nawet nie bardzo wiemy jak obsługiwać dbx. Powód jest prozaiczny: po prostu do tego stopnia zepsuci jesteśmy pięknymi, przyjaznymi i naprawdę porządnymi debuggerami ze środowiska MS Windows (a nawet DOS!), że uczenie się topornego, tekstowego dbx'a wydaje się nam niepotrzebną męką: porównanie dbx'a i debuggera dla (powiedzmy) Visual C++ wypada tak, jak porównanie edytora vi z (powiedzmy) MS Word. Dlatego wolimy poczekać, aż pojawią się porządne debuggery także w Unix'ie (albo, o zgrozo, Windows wyprze i ten system...). Niemniej, osoby chętne, a tym bardziej pragnące pisać duże aplikacje, zachęcamy do wykorzystywania dostępnych debuggerów: jest to inwestycja, która prędzej, czy później, sowicie się opłaci!
Dalej, PETSc dysponuje pewnymi możliwościami graficznymi, ale szczerze powiedziawszy, lepiej nie używać procedur PETSc do wizualizacji: sensowniej jest wykorzystać zewnętrzne programy wizualizacji danych, na przykład AVS, Vigie, czy nawet Matlab. Warto natomiast skorzystać z grafiki PETSc dla obserwacji zachowania własnych obiektów lub procedur. Można w szczególności napisać własne - lepsze! - przeglądarki obiektów itp.
PETSc jest tworzone jako biblioteka otwarta. Jak już wspominaliśmy, PETSc może korzystać z zewnętrznej biblioteki PVODE; jest też port do kilku innych. Między innymi, do bibliotek równoległych BlockSolve95 i SPAI, z dodatkowymi solverami równań liniowych oraz do biblioteki matematycznej IBM ESSL; do Matlaba - dla wizualizacji i ewentualnego postprocessingu numerycznego; do narzędzia wizualizacji trójwymiarowych obiektów przy użyciu VRML (virtual reality), itd.
Najistotniejszą cechą PETSc, która czyni z niej naprawdę poważne narzędzie, jest przejście na programowanie równoległe na wyższym poziomie. To właśnie, w połączeniu z dostępnością podstawowych solverów i narzędzi numerycznych, stanowi prawdziwą smakowitość PETSc! Dlatego zamierzamy w ewentualnej drugiej części tego podręcznika przystępnie wyłożyć generalne zasady programowania równoległego oraz wykorzystania PETSc do (w miarę bezbolesnego) pisania efektywnych aplikacji równoległych, służących rozwiązywaniu równań różniczkowych cząstkowych. Na razie zachęcamy do własnego eksperymentowania z programami równoległymi, wykorzystującymi MPI do uruchamiania jednej aplikacji na kilku stacjach roboczych, co może pozwolić przede wszystkim na rozwiązywanie dużo większych zadań dyskretnych.
Zachęcamy do uczenia się PETSc. To naprawdę szybsze, niż kodowanie wszystkiego od zera...